一、概述
从核磁共振图像(MRI)对整个头部进行精准的分割为个性化的有限元计算模型奠定了基础,提供非侵入性脑刺激(NIBS)等领域的计算机辅助解决方案。由于年龄、遗传史和其他因素,个体头部的结构和功能可能存在很大差异。目前大多数自动头部分割工具都使用健康的年轻人数据开发,因此可能会忽略更容易出现与年龄相关结构性衰退(如脑萎缩)老年人群。
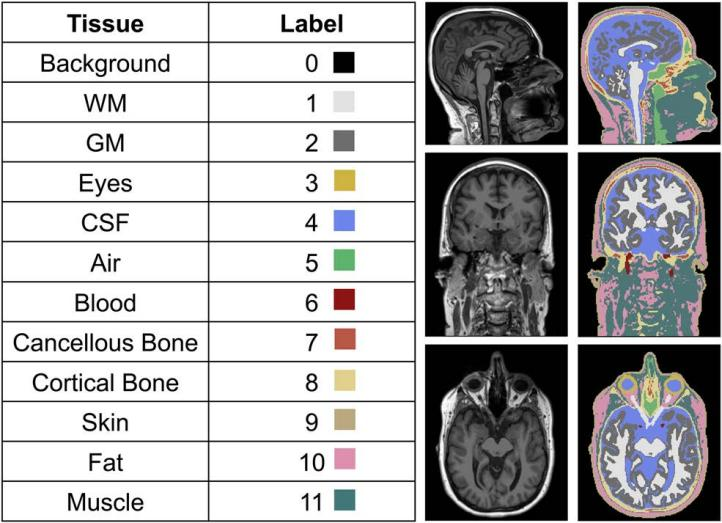
本研究提出一种名为 GRACE 的深度学习方法,在一个新数据集上进行训练和验证,该数据集包含 177 个经过细致人工审查的手动校正 MRI 衍生基准分割。经过训练的 GRACE 模型可从每个 T1 MRI 体积中分割出 11 种组织类型:白质(WM)、灰质(GM)、眼睛、脑脊液(CSF)、空气、血管、松质骨、皮质骨、皮肤、脂肪和肌肉,针对老年人头部进行优化,实现与年龄相关脑部疾病的高精度建模。
二、数据处理
数据集获取
本研究使用的是增强老年人认知训练试验数据,参与的健康老年人群体年龄为 65-89。其中 137 个用于训练,20 个用于验证,20 个用于测试,所有与其他软件的比较都使用相同的测试数据。扫描设备为西门子的 3T Magnetom Prisma 和 3T Magnetom Skyra 磁共振成像断层扫描仪。
基准分割
4 名研究人员使用半自动的标注程序将 MRI 图像数据分割成 11 种组织,这些组织常用于 tDCS 建模。所有自动分割的输出均需要在 Simpleware 软件中进行手动校正。使用 SPM(CAT12) 的 HEADRECO 对 WM、GM 和骨骼进行基础分割,空气在 SPM12 中生成。在 Simpleware ScanIP 中使用阈值工具和形态学操作将骨骼进一步划分为松质骨和皮质骨。脑干、脊髓、视神经、血管、皮肤、脂肪、肌肉和眼睛均在 Simpleware 软件中通过手动分割获得。将包含 11 个组织的掩膜作为训练 GRACE 算法的分割标签,称为“基准分割”。
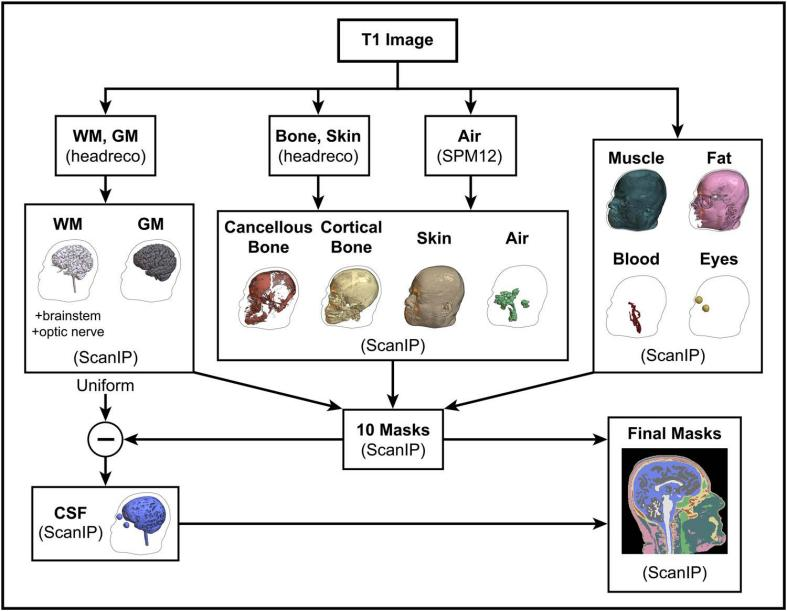

数据前处理
初步阶段将每个 256 × 256 × 176 头部体积随机裁剪成 12 个较小的 64 × 64 × 64 体积扩充训练数据。
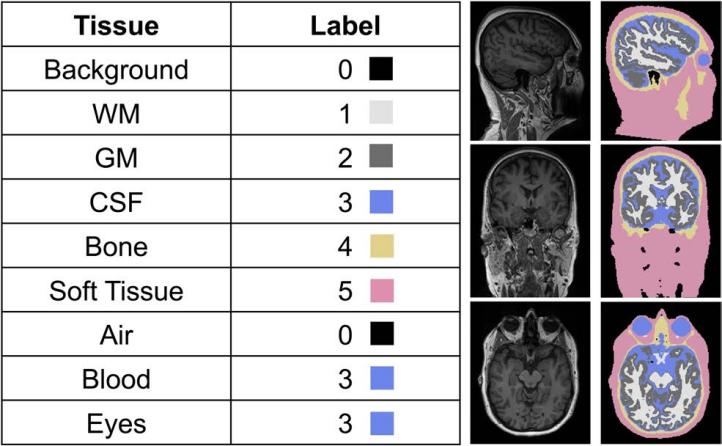
由于不同自动分割方法提供的组织标签不同,为进行比较需要将每种方法的最终组织掩膜合并为更大的类别分组,实现尽可能的公平。
三、GRACE 模型
UNETR 架构
GRACE 采用 UNETR(U-Net Transformer)架构,用 Transformer 模块取代传统 U-Net 网络的编码器路径。UNETR 使用基于全卷积网络(FCN)的解码器,通过跳跃连接将基于 Transformer 的编码器和基于 FCN 的解码器连接起来。
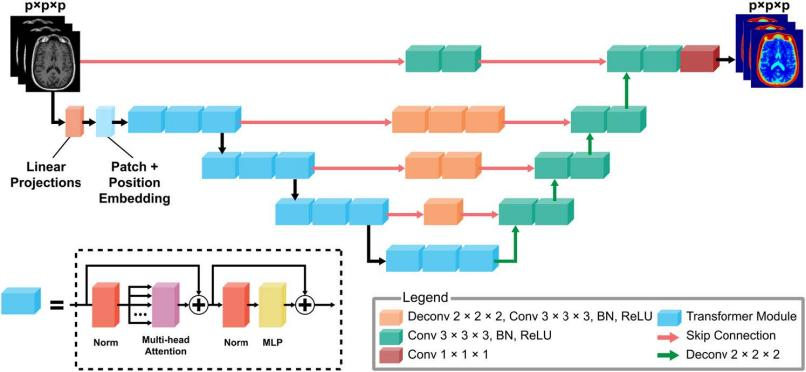
GRACE 和 3D U-Net 均使用由 PyTorch 编写的医学成像深度学习开源 AI 框架 MONAI。GRACE 将 64 × 64 × 64 体积处理为 64 个 16 × 16 × 16 不重叠的体积序列作为输入,U-Net 则使用完整的 64 × 64 × 64 体积。两种模型都对所有网络层随机初始化权重。Adam 优化器以学习率 10-4 和权重衰减 10-5 更新每个模型的参数。GRACE 和 U-Net 分别训练 2500 个周期(epoch),每 50 个周期进行一次验证。训练在超级计算机 HiPerGator 上进行,使用 1 个 A100 NVIDIA GPU、4 个 CPU 和 30 GB RAM。
评估指标
Dice 系数:表示两个二进制掩膜的重叠,可单独计算每个组织的 Dice 系数。数值 1 为完美重叠,0 为完全没有重叠。
平均 Hausdorff 距离:两个数据子集中最近点之间最大距离的平均值。Hausdorff 距离越小,分割效果越好。
四、结果与讨论
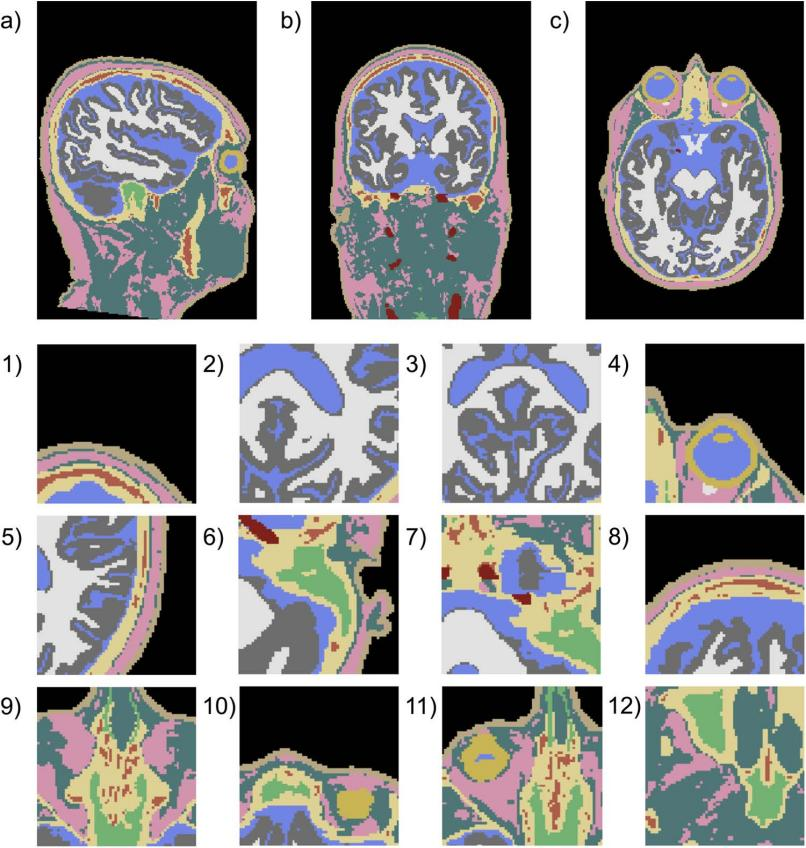
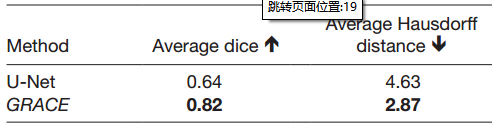
分割 11 个组织
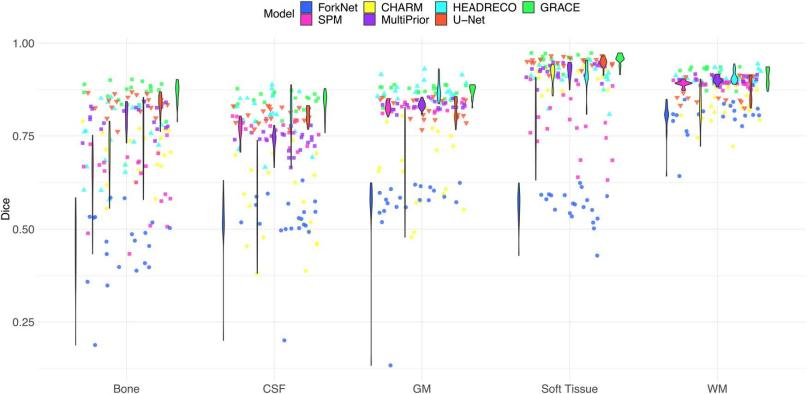
平均 Dice 系数和 Hausdorff 距离表明 GRACE 显著优于 3D U-Net。
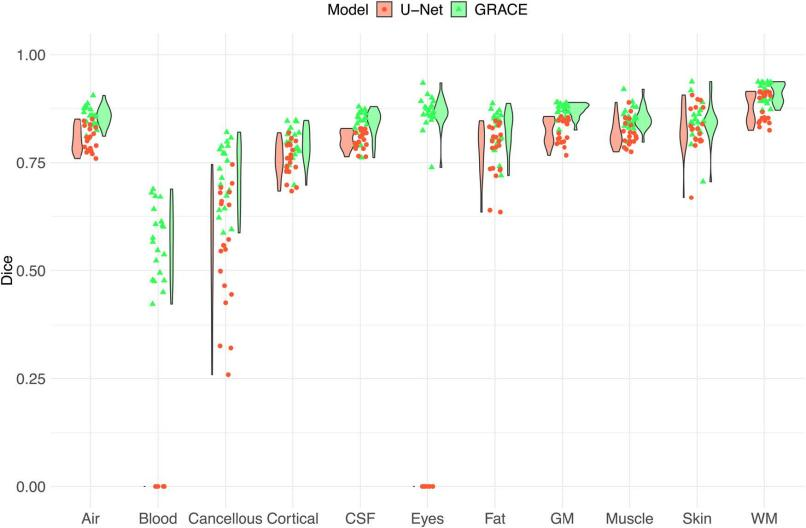
在各种组织中,GRACE 获得的 Dice 系数大致上等于或优于 U-Net,Hausdorff 距离也表现出相同的趋势。特别地,眼睛和血管是 T1 MRI 扫描中体素总数最少的两种组织,U-Net 根本无法识别出。
分割 5 个组织
为进行比较,将组织合并为更大的标签类别。结果表明,GRACE 展现出最高的 Dice 系数和最低的 Hausdorff 距离,即 GRACE 的平均表现优于其他每种方法。
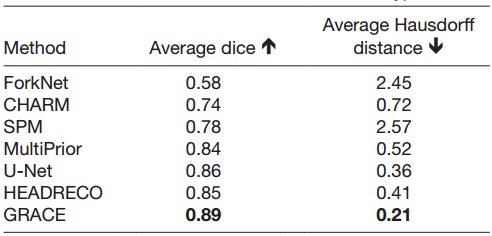
GRACE 在 CSF、骨骼和软组织方面获得最高的 Dice 分数,WM 和 GM 组织的 Dice 分数与 HEADRECO 相当。Hausdorff 距离也表现出相同的趋势。
定性结果
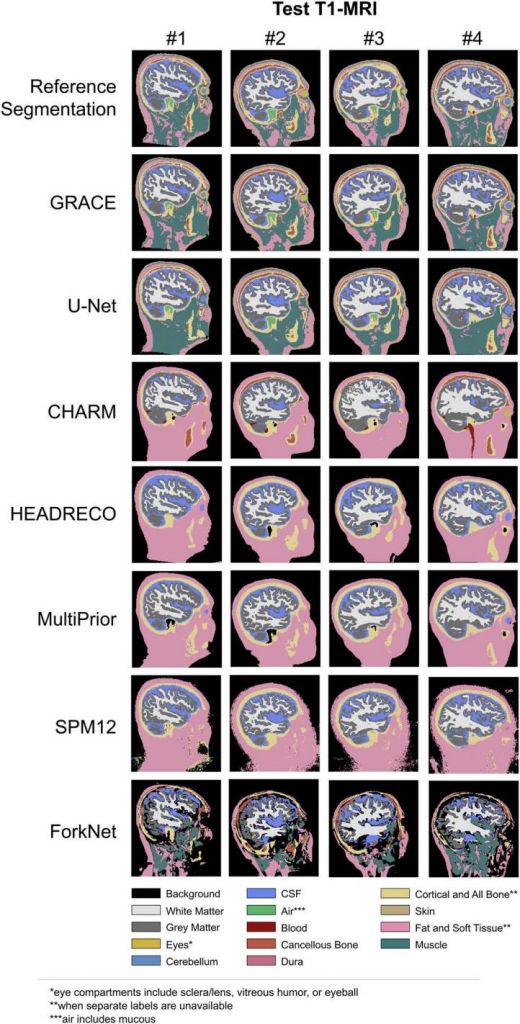
- SPM12 结果中皮肤边界周围很嘈杂,一些背景中的体素也被分配给软组织。
- CHARM 最初在 #1 测试数据上产生了较差的配准结果,需要运行两次修复仿射配准,使分割时间加倍。在所有测试中都缺失了颅腔中的大量 CSF,但它可以很好地分割眼睛。
- HEADRECO 在区分头部形状、WM 和 GM 方面是免费软件工具中最好的,在骨骼上较差。
- MultiPrior 与 HEADRECO 相似,但缺失一些关键细节,眼睛和下巴都被分割为较薄的结构。
- U-Net 产生的结果与 GRACE 有些相似,也仍缺乏重要的细节,如松质骨、眼睛、脂肪等。
- ForkNet 尝试标记许多类别的组织,但实现上比较困难。它将头部的很大一部分识别为背景,尤其是难以处理的脸部前方,还将大量肌肉置于颅腔内。
- GRACE 在 #3 中缺失了眼睛,但它是唯一在 #2 中正确区分出眼睛的分割工具。GRACE 在对眼睛、松质骨、皮质骨、皮肤、脂肪和肌肉的精细分割方面特别有优势。关于 WM、GM 和 CSF 则与HEADRECO 相当。
改进基准分割
GRACE 的另一个优势是它有潜力改进具有挑战性的基准分割。当唯一可用成像方式为 T1 MRI 时,血管是最难识别和标记的组织之一。初步定性结果表明,与基准分割搭配使用时,GRACE 可以提高血管分割的质量。第一次运行的 GRACE 分割可以识别出通常出现在大脑区域附近的动脉前端,这个特定区域很难通过人眼与 T1-MRI 区分。基准分割中的血管经过校正后,GRACE 会使用相同的数据集重新训练,提高血管分割的准确性。
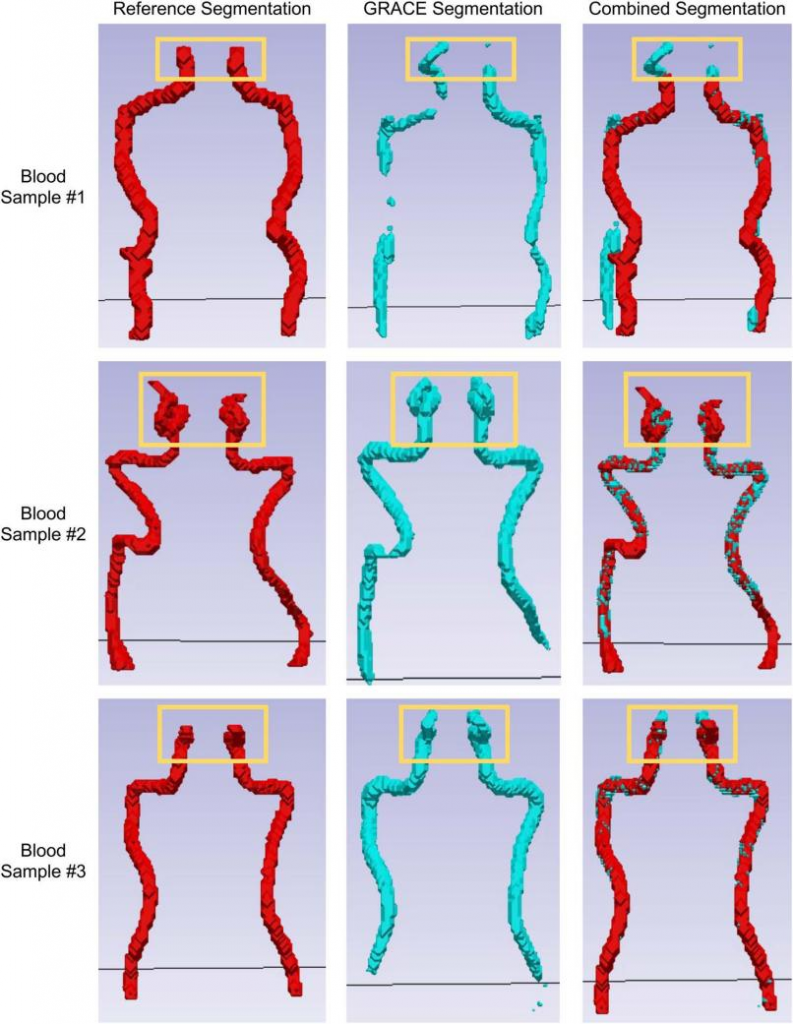
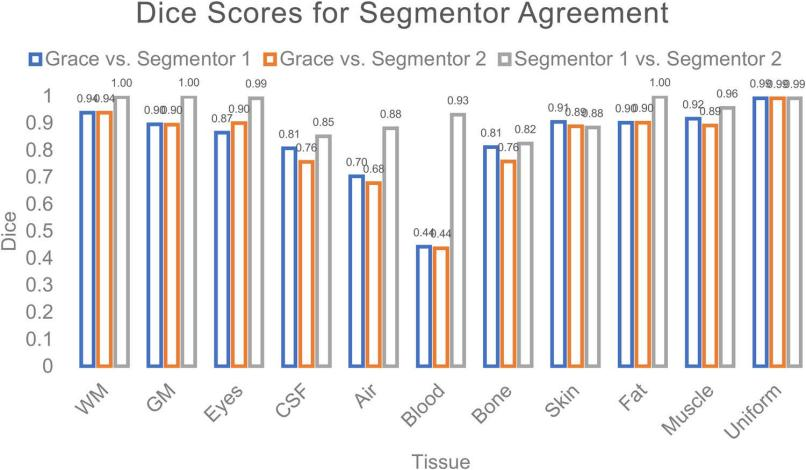
分割一致性
将 GRACE 与研究人员的手动分割结果进行比较。所有研究人员的手动分割结果重叠 Dice 系数都在 0.79-0.99,GRACE 与研究人员手动分割重叠度在很大程度上也算相当一致。具有更复杂的组织几何形状和更少体素数量(如血管)的组织是 GRACE 未来的改进方向。
时间计算成本
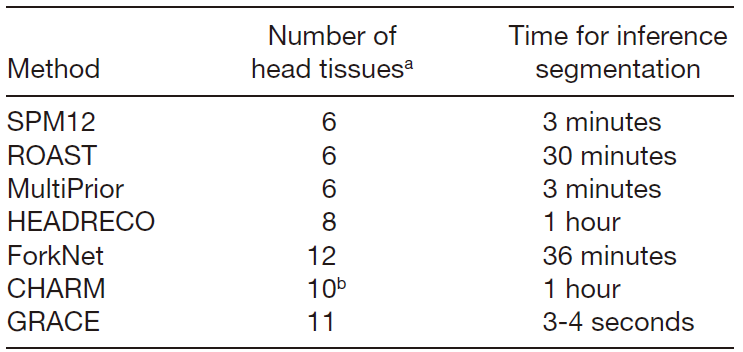
比较使用不同软件完成 176 × 256 × 256 T1 MRI 图像体积分割的平均时间,GRACE 耗时最短,仅需 3-4 s。
五、结论
本研究提出了一种从 T1 MRI 扫描图像数据中自动分割出 11 种不同头部组织的新方法 GRACE。与其他六种免费工具(CHARM、HEADRECO、SPM、U-Net、ForkNet、MultiPrior)相比,GRACE 更具优势。GRACE 在传统上具有挑战性的组织中实现了相对较高的准确度和效率,包括与老年人群体相关的组织(如脑萎缩和骨质疏松症)。深度学习的主干架构为新分割任务和具有较小数据集的不同人群提供了灵活性和可扩展性。
六、参考
- Stolte S E, Indahlastari A, Chen J, et al. Precise and rapid whole-head segmentation from magnetic resonance images of older adults using deep learning[J]. Imaging Neuroscience, 2024, 2: 1-21.
- GRACE 代码:https://github.com/lab-smile/GRACE
