目录
寻找分子真实构型/多个异构体共存的分子的谱学性质的玻尔兹曼平均、生成*.coskf文件
工作逻辑
- 力场计算,不同构型的能量,误差很大。因此能量大小顺序可能也有误差。但是速度极快。
- DFTB 或 Mopac,计算能量误差较小,能量大小顺序基本上正确。速度略慢。
- ADF 计算能量很准确,但是速度很慢。因此考虑到效率和可靠性的综合,力场适合大范围粗筛,ADF 适合最后精准筛选。如果一开始就用 ADF 精筛,速度将会慢到无法接受。
注意,以上流程将使用ADF、DFTB、ML Pot模块。
工作流程概述
- 用户创建原子的连接关系(原子之间设置正确键级),由 Conformer 功能找出所有可能的三维分子结构
- 进一步略微提高精度(使用 Mopac),计算所有结构的能量,然后根据设定的 Max energy,去掉比能量最低构型高出 Max energy 以上的那些构型
- 再次提高精度(使用 ADF),随着计算能量的方法的精度提高,Max energy 可以设置的比前面小,例如 1 kcal/mol,从而保留的能量最低的结构更少,最后只剩下能量最低的 1~3 个,即正确的三维构型
- 如果存在多个能量最低的构型,针对多个构型计算光谱性质,或生成*.coskf文件
如果用户只是想找出真实构型,则“第三步:使用ADF计算多个异构体的热力学平均光谱(玻尔兹曼平均)”这一步不需要做。在前面的最后步骤完成后,在 SCM → Movie 中查看能量最低的三维构型,即可用于其他场景的使用。
第一步:创建初步分子结构
1.1 建模与参数设置
分子建模教程参考:建模
建模要求:不要求键长键角正确,但键级需要正确设置。
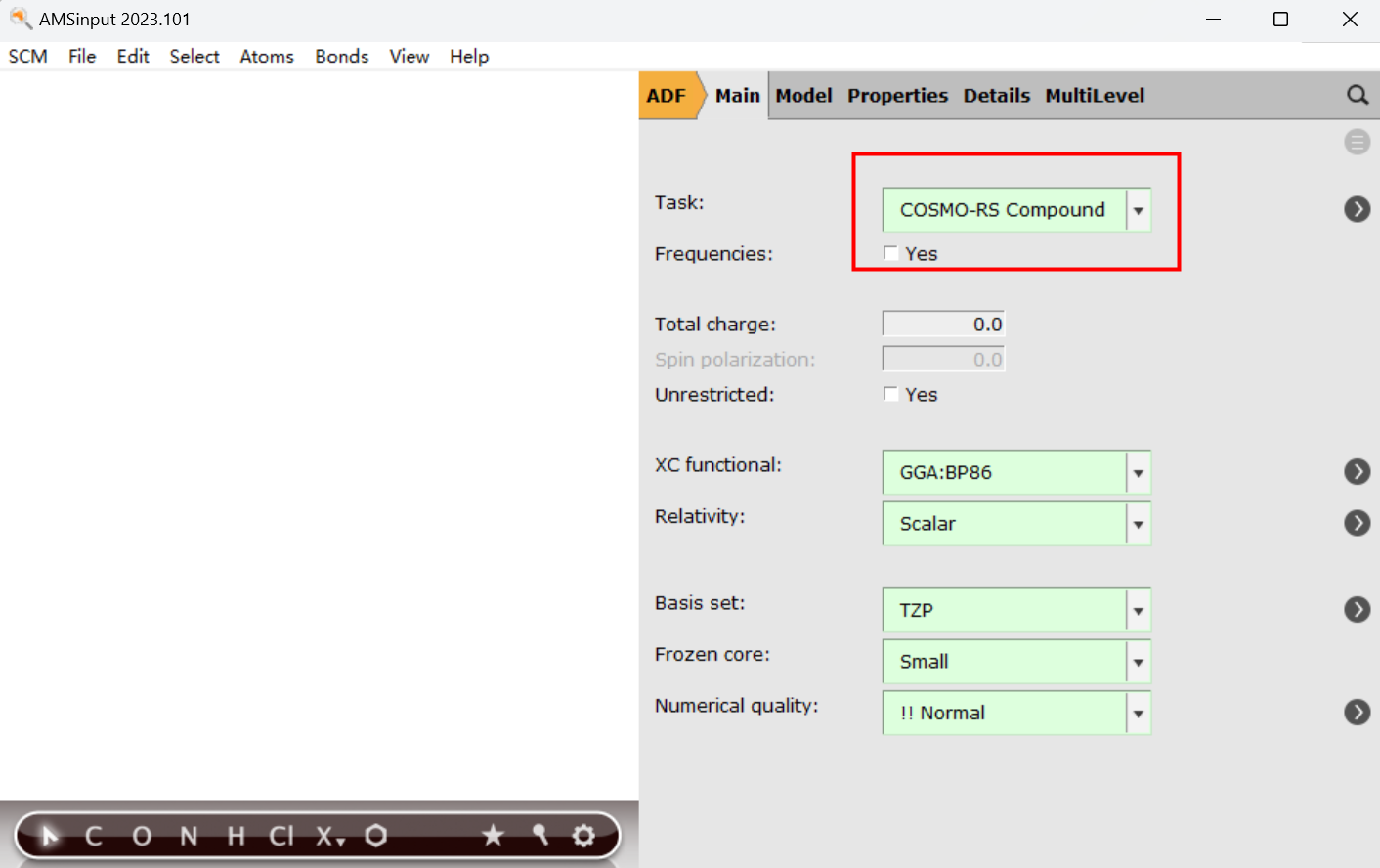
参数设置如上图所示(切换到Conformer模块,均使用默认参数即可)。其中Max Energy指能量最低的一个范围的构型,其中能量是由指定的计算引擎优化结构后得到,例如这里指定ForceField(默认使用UFF力场)。超过最低能量 4 kcal/mol 的结构将被滤除。因为UFF力场一般倾向于低估不同构型的能量差值,因此调用UFF时,Max energy不建议设置太大,分子如果比较小,设置为 3kcal/mol 更高效。
如果是大分子,则建议关闭GCStep以节省时间:
File - Save保存任务,并运行。
1.2 计算过程日志
SCM → logfile:
[23.04|13:47:38] Conformers 2023.101, Nodes: 1, Procs: 1 [23.04|13:47:38] Molecule: C4H10O2, 16 atoms, 3 rotatable bonds [23.04|13:47:39] Generating 2700 conformers with RDKit ... [23.04|13:47:42] RDKit conformer generation finished! Time taken: 0:00:03s [23.04|13:47:42] Running 1000 geometry optimizations with 1 worker [23.04|13:48:42] 405 / 1000 jobs finished: 40.5%, 0:01:28s remaining [23.04|13:49:42] 741 / 1000 jobs finished: 74.1%, 0:00:42s remaining [23.04|13:50:28] All 1000 geometry optimizations done! Time taken: 0:02:45s [23.04|13:50:30] Running 1000 geometry optimizations with 1 worker [23.04|13:51:30] 383 / 1000 jobs finished: 38.3%, 0:01:37s remaining [23.04|13:52:30] 791 / 1000 jobs finished: 79.1%, 0:00:32s remaining [23.04|13:53:06] All 1000 geometry optimizations done! Time taken: 0:02:36s [23.04|13:53:08] Running 700 geometry optimizations with 1 worker [23.04|13:54:08] 442 / 700 jobs finished: 63.1%, 0:00:35s remaining [23.04|13:54:41] All 700 geometry optimizations done! Time taken: 0:01:33s [23.04|13:54:41] 2700 out of 2700 geometry optimizations succeeded [23.04|13:54:41] Filtering 2700 conformers [23.04|13:54:49] Conformer filtering complete! Time taken: 0:00:07s [23.04|13:54:49] 2686 conformers filtered out as duplicates [23.04|13:54:49] 0 high energy conformers filtered out [23.04|13:54:49] Remaining number of conformers in set is 15 [23.04|13:54:49] Final duplicate filtration [23.04|13:54:49] Filtering 15 conformers [23.04|13:54:49] Conformer filtering complete! Time taken: 0:00:00s [23.04|13:54:49] 0 conformers filtered out as duplicates [23.04|13:54:49] 0 high energy conformers filtered out [23.04|13:54:49] 0 high energy conformers filtered out [23.04|13:54:49] Found 15 unique conformers. Job conformer01 has finished
这里显示的信息包括:
- 使用1个核心进行计算耗时7分11秒,如果使用8核,耗时约1分钟
- 分子式: C4H10O2, 16 个原子, 3 可以转动的键
- 产生了2700个结构
- 优化其中1000个结构
- 优化另外1000个结构
- 优化另外700个结构
- 排除了2686个等价结构只剩下15个不等价结构(此处仍然有少量等价结构,可以通过后面的筛选过程进一步自动筛除)
1.3 结果查看
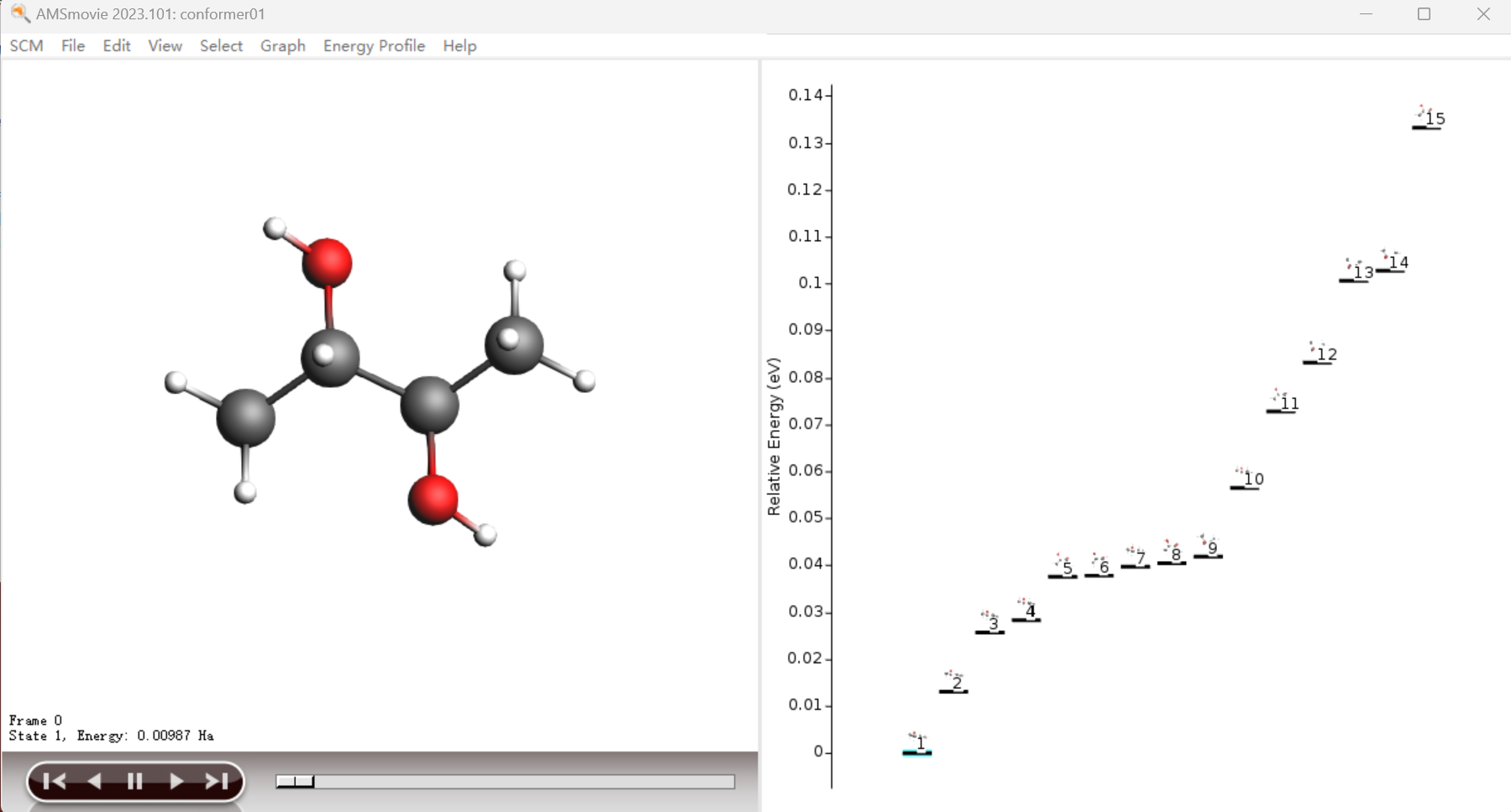
任务结束后,SCM - Movie可以看到搜索结果,得到15个异构体(左边窗口是三维构型,右边窗口显示各种构型的能量):
当然能量最低的是第一个结构。使用键盘的← →按键,可以查看各种异构体。这些搜索得到的结构,都已经使用UFF力场优化过了,对应的能量是用UFF力场计算得到的。默认能量单位为 Hatree,双击纵坐标,可以修改能量单位,例如改为 kcal/mol。
第二步:逐步提高精度,依次使用Mopac、ADF继续筛选结构
我们可以仍然在Conformer中进行筛选,基于前面得到的15个结构,采用更高精度的计算引擎例如MOPAC进一步滤除高能量构型。
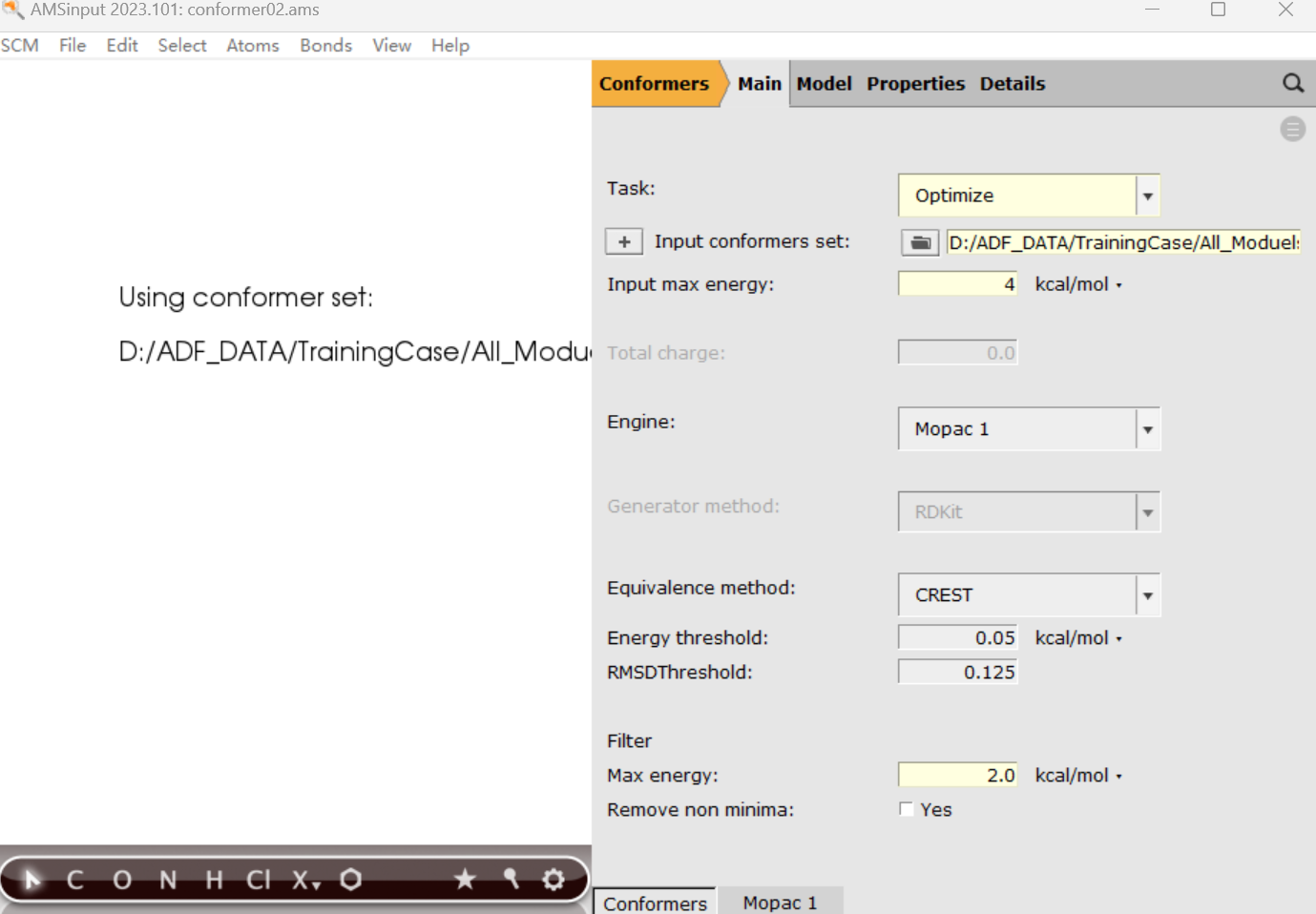
2.1 Mopac 筛选的参数设置
- Task选择Optimize,表示对前面得到的15个结构进一步筛选
- Input conformers set选择前一个作业生成的*.results/conformers.xyz文件
- Input max energy这里表示只读入上次计算生成的15个结构中,能量从低到高,范围在4 kcal/mol以内的那些结构,conformers.xyz文件中有每个结构对应的能量值。如果前一步生成的结构非常多,这里可以可以考虑降低Input max energy,从而少输入一些结构,供本轮筛选。
- Engin改为Mopac,Mopac默认使用PM7方法计算,这个一般不需要修改
- Max energy设置的较低1.5kcal/mol,即Mopac计算这15个结构,能量从低到高排序,只输出范围在2.0kcal/mol的这些少量结构。
2.2 Mopac 筛选结果查看
SCM → Movie:
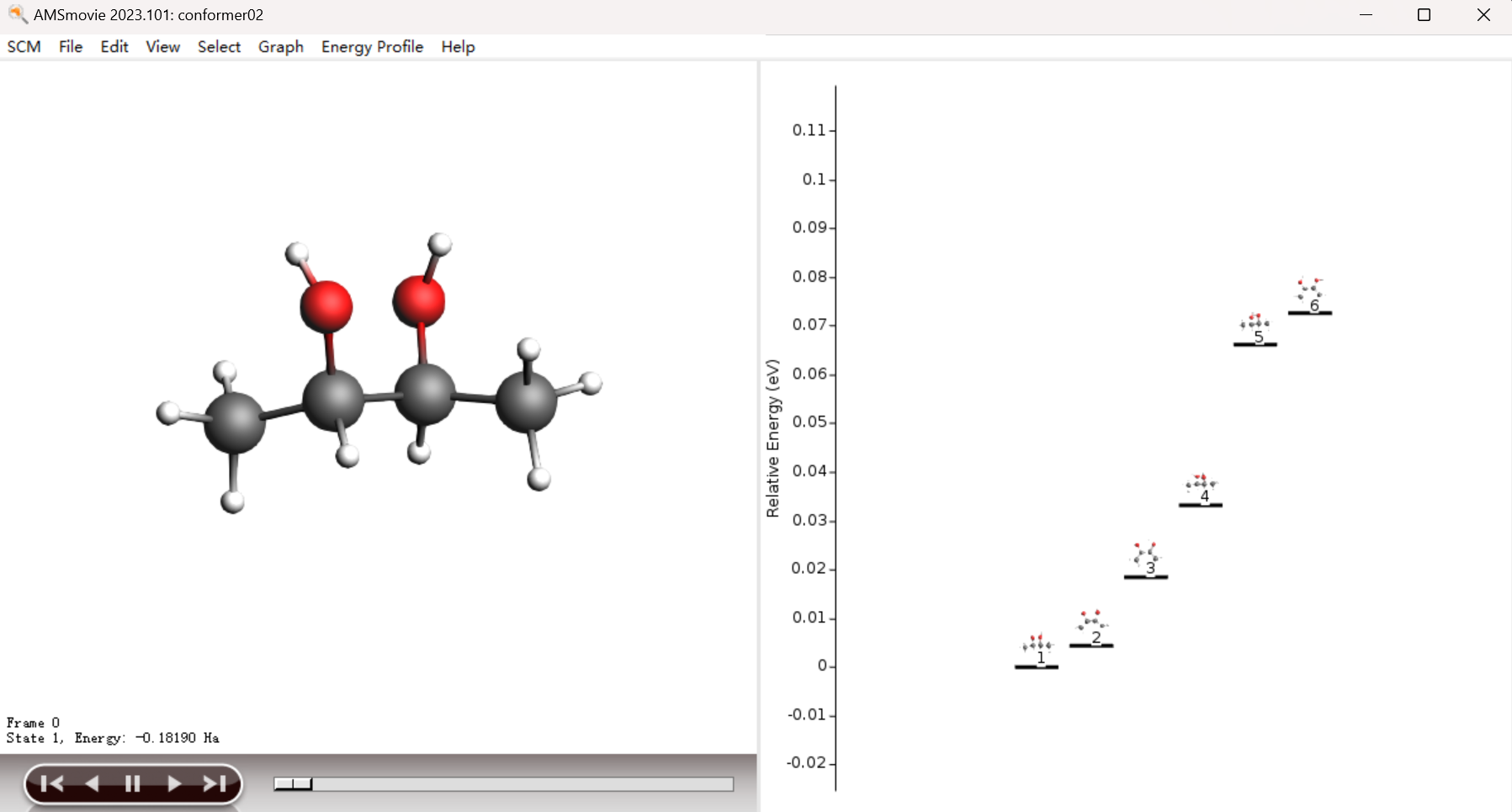 因为前面设置了“只输出范围在2.0kcal/mol的这些少量结构”,计算结果中保留了符合这个要求的6个结构。这些能量的数值是用 Mopac 计算得到的,数值的准确性、大小顺序的可靠性比力场高很多。
因为前面设置了“只输出范围在2.0kcal/mol的这些少量结构”,计算结果中保留了符合这个要求的6个结构。这些能量的数值是用 Mopac 计算得到的,数值的准确性、大小顺序的可靠性比力场高很多。
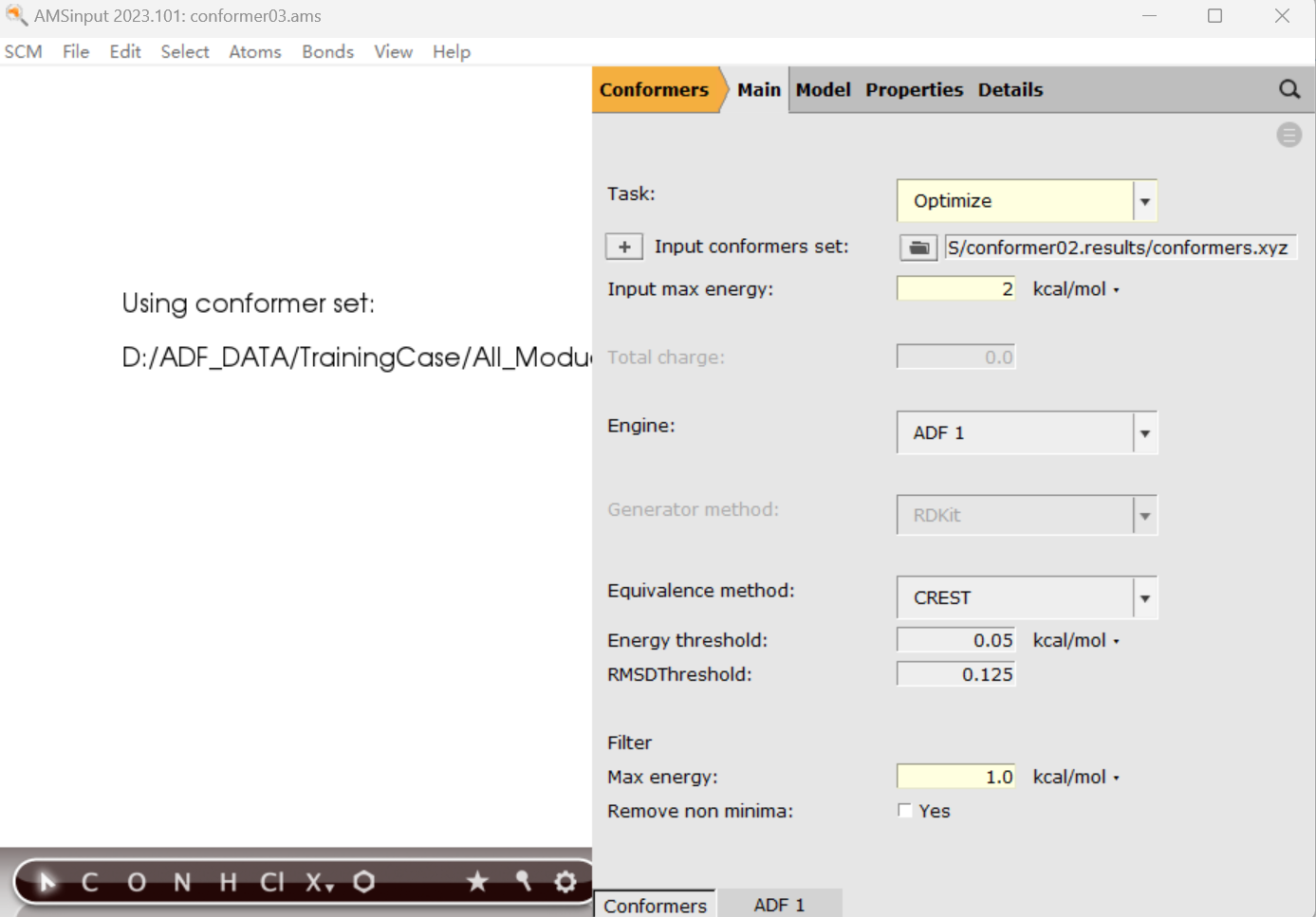
2.3 ADF 筛选的参数设置
ADF 模块(参数如下图所示,为 DZP 基组、BP86 泛函、相对论-None,Max energy 为 1 kcal/mol) 精度比 Mopac 高,如此进一步精细筛选。
筛选过程,甚至给出了预估时间和进度:
[23.04|16:01:44] Conformers 2023.101, Nodes: 1, Procs: 6 [23.04|16:01:44] Molecule: C4H10O2, 16 atoms, 3 rotatable bonds [23.04|16:01:44] Reading conformers from file: D:/ADF_DATA/conformer02.results/conformers.xyz [23.04|16:01:44] 0 high energy conformers filtered out [23.04|16:01:44] 6 conformers read from file. [23.04|16:01:44] Running 6 geometry optimizations with 6 workers [23.04|16:02:44] 0 / 6 jobs finished: 0.0% [23.04|16:03:44] 0 / 6 jobs finished: 0.0% [23.04|16:04:44] 0 / 6 jobs finished: 0.0% [23.04|16:05:44] 0 / 6 jobs finished: 0.0% [23.04|16:06:44] 2 / 6 jobs finished: 33.3%, 0:10:00s remaining [23.04|16:07:44] 4 / 6 jobs finished: 66.7%, 0:03:00s remaining [23.04|16:08:44] 4 / 6 jobs finished: 66.7%, 0:03:30s remaining [23.04|16:09:44] 4 / 6 jobs finished: 66.7%, 0:04:00s remaining [23.04|16:10:44] 5 / 6 jobs finished: 83.3%, 0:01:48s remaining [23.04|16:11:44] 5 / 6 jobs finished: 83.3%, 0:02:00s remaining [23.04|16:12:44] 5 / 6 jobs finished: 83.3%, 0:02:12s remaining
2.4 ADF 筛选结果查看
计算完毕,仍可如前一样通过SCM → Movie查看不同构型的能量,选择能量最低的构型(这些能量是由高精度方法 ADF 生成,能量的数值、大小顺序都极为可靠),然后菜单栏File → Export Coordinates将该构型保存为xyz文件,即用于其他场景,例如生成*.coskf文件。
第三步:使用ADF计算多个异构体的热力学平均光谱(玻尔兹曼平均)
这里是选择性的第三步。
3.1 结构导入与参数设置
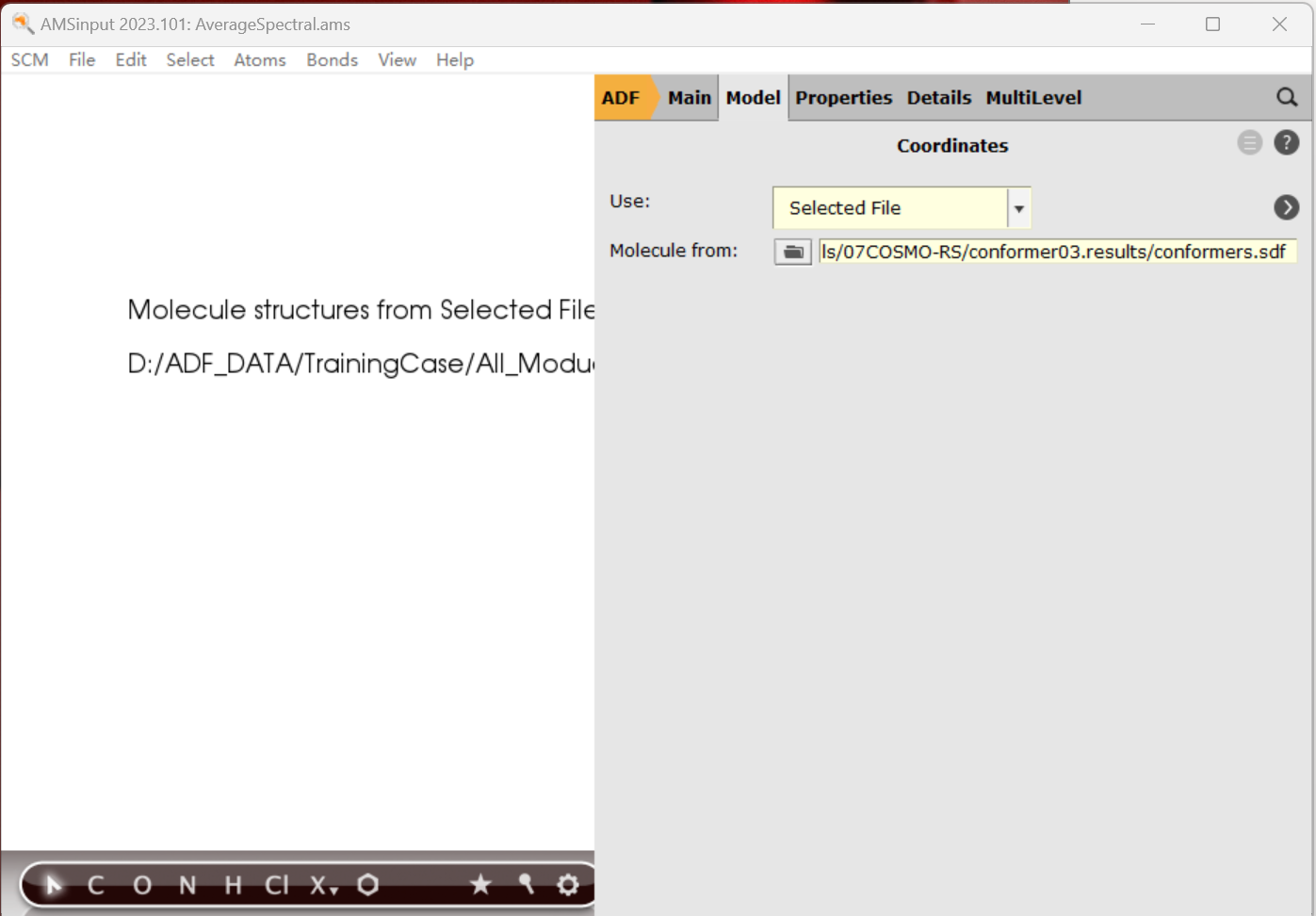 此处读入最后一步结构筛选生成的*.results/conmformers.sdf文件。
此处读入最后一步结构筛选生成的*.results/conmformers.sdf文件。
由于最后一次筛选可能还剩下5个结构或更多,这里我们尝试只计算能量最低的3个结构的红外光谱,因此这里填入数字3:
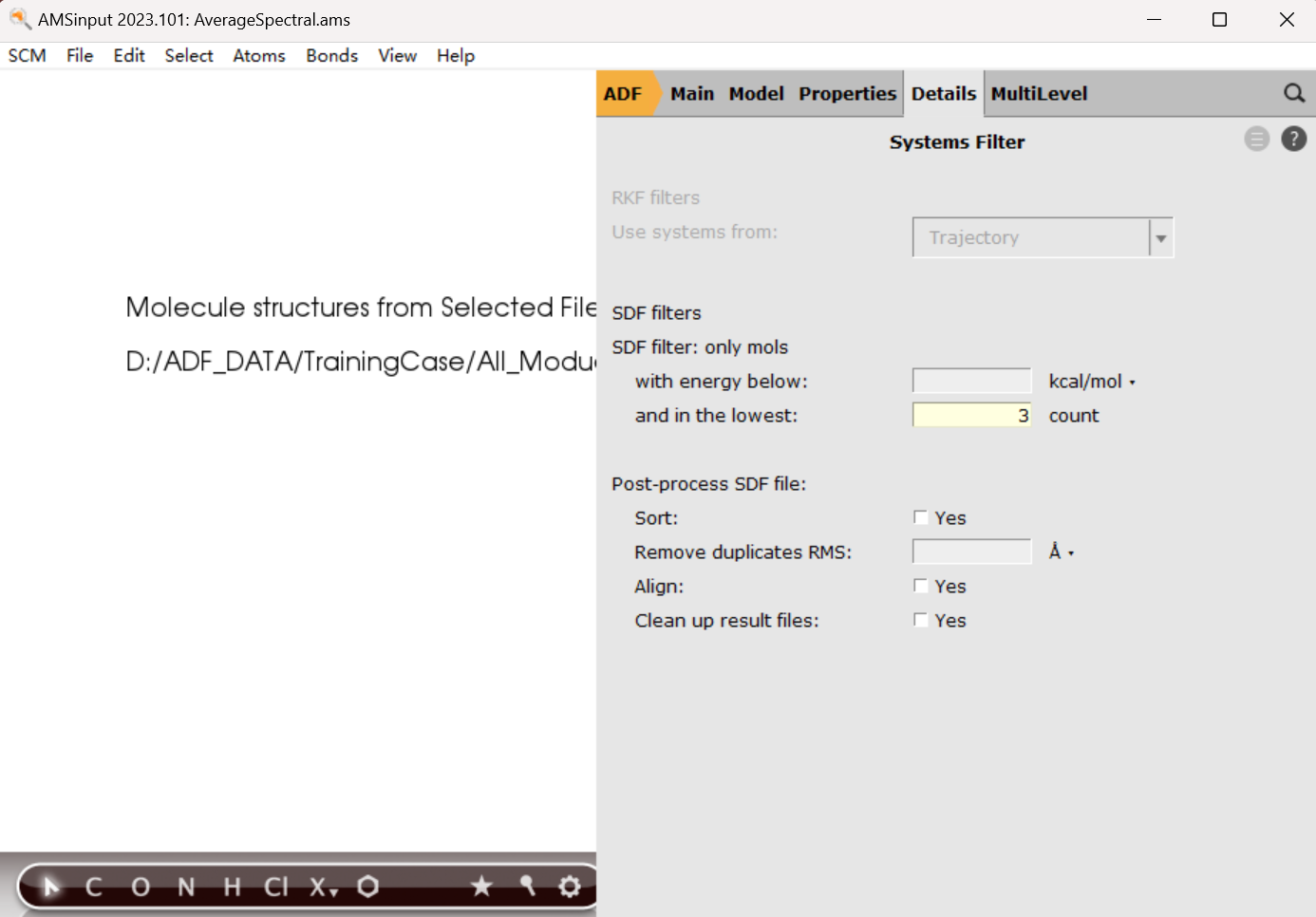
设置红外光谱计算的参数:
 当然用户可以使用精度更高的泛函、基组等。不仅仅是红外光谱的计算可以做玻尔兹曼平均,紫外光谱等也都可以。此处设置紫外光谱等性质的参数即可。
当然用户可以使用精度更高的泛函、基组等。不仅仅是红外光谱的计算可以做玻尔兹曼平均,紫外光谱等也都可以。此处设置紫外光谱等性质的参数即可。
保存并运行作业。
3.2 结果查看
SCM → Spectra:
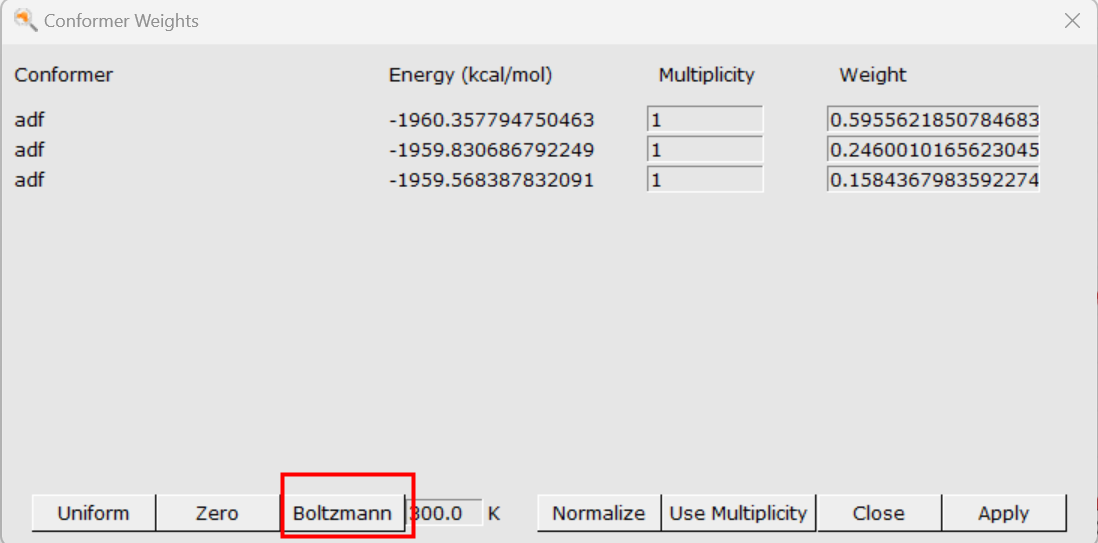
该谱图即为平均后的结果(温度为300K,其他温度下的均值,在此处直接修改温度即可)。点击谱图右下角的Boltzmann下拉菜单选择Uer:
点击Boltzmann即可显示三种异构体在300K下的占比。用户可以调整不同温度,得到不同温度下的占比,以及对应的谱图。