缺陷碳纳米管的杨氏模量
碳纳米管以其异常的机械性质而著称;被测得有1TPa量级的杨氏模量[KDE+98],高于所有已知材料。对于无缺陷的碳纳米管杨氏模量的理论计算相当简单,但是当有缺陷存在时就另当别论了。对此,分子动力学(MD)是计算机械性能的合适工具。 在本实例中你将构造具有Stone–Wales缺陷的碳纳米管(CNT),并使用ATK-Classical来计算杨氏模量。
弹性模量
杨氏模量是指材料在弹性区应力$\sigma$和应变$\varepsilon$的关系,满足胡克定律:$E = \sigma / \varepsilon$
重要提示!
本实例使用在实例Stone–Wales defects in nanotubes中构造的CNT结构。如果你还没有那样做,请先运行那个实例并构建CNT器件构象,或者直接下载Python脚本:stone_wales_cnt.py。
CNT块体构象
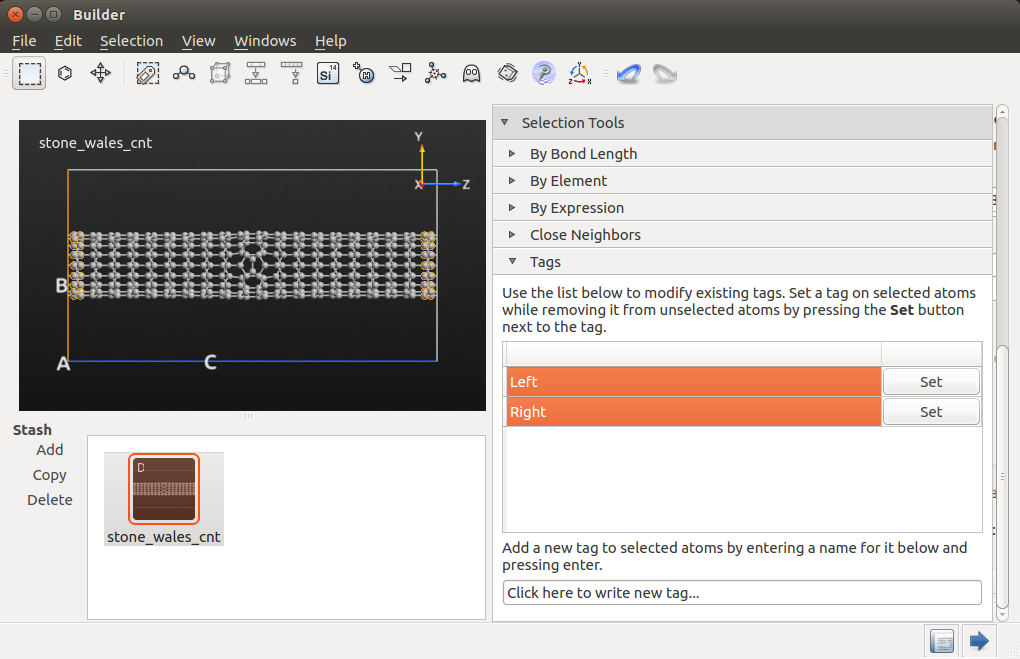
打开VNL Builder![]() , 使用 File ‣ Add ‣ From Files将之前保存的CNT构象导入Stash。注意到这个结构是一个中间区域有Stone–Wales缺陷的器件构象。
, 使用 File ‣ Add ‣ From Files将之前保存的CNT构象导入Stash。注意到这个结构是一个中间区域有Stone–Wales缺陷的器件构象。
接下来,选中器件的Stash项(显示为高亮),点击![]() 按钮将其转化为块体构象(只是将器件电极移除),并使用Selection Tools ‣ Tags工具将纳米管两端的原子添加标签:
按钮将其转化为块体构象(只是将器件电极移除),并使用Selection Tools ‣ Tags工具将纳米管两端的原子添加标签:
- 使用鼠标将最左端的碳原子选中(显示为高亮)并将它们贴上标签“Left”。
- 对最右端的碳原子做同样的操作,如下图所示。
这些标签将会被用来在分子动力学(MD)模拟中对结构施加约束。
为分子动力学模拟创建构象
将带有Stone–Wales缺陷的CNT块体构象送到Script Generator![]() 并添加 New Calculator
并添加 New Calculator![]() 和
和![]() Optimization ‣ MolecularDynamics 模块。然后编辑各个模块来对计算进行设置:
Optimization ‣ MolecularDynamics 模块。然后编辑各个模块来对计算进行设置:
- 首先,将默认输出文件改为mdtrajectory_cnt.nc。
- 在 New Calculation模块中选择ATK-Classical 计算器,并确保使用 Tersoff_C_2010 经典势(为模拟石墨烯和纳米管晶格动力学而设计的势)。
- 在MolecularDynamics模块中进行以下修改:
Molecular Dynamics选项:
- 设置 “Type” 为 NVT Berendsen。
- 设置“Steps” 为 20000。
- 设置“Log interval”为 200。
Initial Velocity选项:
- 设置“Type” 为Maxwell-Boltzmann。
- 设置“Temperature” 为300 K。
- 点掉 Remove center-of-mass momentum 勾选项。
NVT Berendsen选项:
- 设置“Thermal coupling”为 1 fs。
如上图所示,保持其他设置为默认值。
添加Python挂钩
在QuantumATK中可以定义“挂钩”;即在每个MD步之前或之后被触发的Python函数或类。这些挂钩可被用来提供自定义的实时分析或者监测系统,或者-如本实例-在模拟的过程中动态地修改结构。
你现在将定义一个预处理挂钩用以在每一步分子动力学模拟中给纳米管一个小的拉伸:
- 在Editor中打开Python脚本,在脚本底部找到定义md_trajectory的模块。之前定义的“Left”和 “Right” 标签现在可以被用来对系统添加约束。改变语句
constraints=[]
为
constraints=bulk_configuration.indicesFromTags(["Left","Right"])
- 在 md_trajectory 模块添加语句
pre_step_hook=strainer,
并在它紧前面的语句添加一个逗号(method=method,)。
- 你也应当定义什么是“strainer”:在 md_trajectory 模块紧前面添加以下语句:
strainer = StressStrainUtility()
- 脚本底部现在应该如这样:
# ------------------------------------------------------------- # Molecular Dynamics # ------------------------------------------------------------- initial_velocity = MaxwellBoltzmannDistribution( temperature=300.0*Kelvin, remove_center_of_mass_momentum=False ) method = NVTBerendsen( time_step=1*femtoSecond, reservoir_temperature=300*Kelvin, thermal_coupling_constant=1*femtoSecond, initial_velocity=initial_velocity ) strainer = StressStrainUtility() md_trajectory = MolecularDynamics( bulk_configuration, constraints=bulk_configuration.indicesFromTags(["Left","Right"]), trajectory_filename='trajectory.nc', steps=20000, log_interval=200, method=method, pre_step_hook=strainer, ) bulk_configuration = md_trajectory.lastImage()
- 上图的17行初始化了类 StressStrainUtility的一个实例,现在你将定义这个Python类。在脚本最顶部嵌入以下语句:
# Define a prestep function which can expand the system class StressStrainUtility: """ Perform a strain of a configuration over time. """ def __init__(self, delta=0.0001, skip=1000): """ @param delta The strain to apply in each step @param first steps to "skip" strain """ self.__delta = [0.0,0.0,delta]*Angstrom self.__skip = skip self.stresses = [] self.lengths = [] def __call__(self, step, time, configuration): """ Strainer call. """ # Get the lattice vectors. u1, u2, u3 = configuration.bravaisLattice().primitiveVectors() # Don't apply strain in the earliest MD steps # if step < self.__skip: # return # Calculate the stress. stress = Stress(configuration) # Store the history self.stresses.append( stress.evaluate()[2,2].inUnitsOf(eV/Angstrom**3) ) self.lengths.append( u3[2].inUnitsOf(Angstrom) ) # Increase the lattice vectors. u3 = u3 + self.__delta # Set the new lattice. configuration._setBravaisLattice(UnitCell(u1,u2,u3)) # Move all the atoms that are constrainted on the right side. constraints = configuration.indicesFromTags("Right") for c in constraints: xyz = numpy.array(configuration.cartesianCoordinates()[c]) + numpy.array(self.__delta) configuration._changeAtoms(indices=[c], positions=[xyz]*Angstrom) def length(self): return numpy.array(self.lengths)*Angstrom def stress(self): return numpy.array(self.stresses)*eV/Angstrom**3
这个类定义了一个可以对单胞沿z方向拉伸的方法。你将使用它来研究一个有缺陷的CNT如何对应变做出响应。 正如你在脚本中所见,这个类有两个输出,分别是delta 和skip:
delta 定义了每一步MD模拟中u3矢量(z方向)扩展了多少。
skip 定义了在构象被拉之前经过多少步。
实际的拉伸是通过41行起的“for-loop” 完成的,沿着z方向以delta 值增加被标记为“Right”的原子的坐标。在30和31行,计算应力和构象总长度
并分开地列着。这将在之后的绘图中用到。
提示!
通过在md_trajectory 模块中写入pre_strain_hook=strainer,这个类(严格的说是call类函数)在每一个MD步之前被调用。
计算杨氏模量
为了正确地计算杨氏模量,有必要考虑模拟域的总体积。QuantumATK得出的应力必须乘以晶胞的总体积然后除以纳米管的体积。 有几种定义CNT体积的方法。一个通常的定义方法是说纳米管壁在管壁处向内外分别伸出半个$\pi$键长,所以它是一个具有厚度等于$\pi$键长(约为1.35Å)的圆柱壳,也就是说横截面积为1.35Å乘以纳米管的周长。一个(5,5)-armchair纳米管的周长为$5 \times (3 \times x)$,x=1.42Å是碳碳键长。最后,为了求体积,你需要管的长度,在QuantumATK保存的 .nc 文件中给出。
- 在脚本底部,删除语句
nlsave('mdtrajectory_cnt.nc', md_trajectory)
- 用以下语句代替来调用恰当的应力计算:
# ------------------------------------------------------------- # Finding Young's Modulus # ------------------------------------------------------------- # Find center of cnt from mean x- and y-coordinates coords = bulk_configuration.cartesianCoordinates() x_coords = coords[:,0] y_coords = coords[:,1] x_0 = numpy.sum(x_coords)/len(x_coords) y_0 = numpy.sum(y_coords)/len(y_coords) # Find radius by mean distance from center to atoms' x- and y-coordinates radius = numpy.sum( ( (x_coords - x_0)**2 + (y_coords - y_0)**2 )**(1/2.0) ) / len(x_coords) # Find cross-sectional area by multiplying circumference with length of a pi-bond pi_bond = 1.35 circumference = radius * 2 * numpy.pi area = circumference * pi_bond # Find volumes of CNT and of whole cell volume_cnt = area * strainer.lengths[-1] u1, u2, u3 = bulk_configuration.bravaisLattice().primitiveVectors() volume_cell = u1[0] * u2[1] * u3[2] # Factor the stresses to the volume of the CNT stresses_factored = numpy.array(strainer.stresses * (volume_cell / volume_cnt) )*eV/Angstrom**3 # Calculate the strain strain = (strainer.length() - strainer.length()[0] ) / strainer.length()[0] # Calculate Young's modulus in TPa print "Young's Modulus: ", (stresses_factored[-1].inUnitsOf(Pa) - stresses_factored[0].inUnitsOf(Pa) ) / float(strain[-1]) / 1e12, " TPa"
- 最后,添加几行语句以保存计算的不同部分并用pylab绘出结果。
# Save the result to a file. print strainer.length() nlsave("mdtrajectory_cnt.nc", md_trajectory, "MD") nlsave("mdtrajectory_cnt.nc",strainer.length(), "C length") nlsave("mdtrajectory_cnt.nc",strainer.stress(), "C stress") nlsave("mdtrajectory_cnt.nc",stresses_factored, "C stresses factored") # Plot stress-strain curve. import pylab pylab.figure() pylab.plot(strain * 100, stresses_factored.inUnitsOf(GPa)) pylab.title('CNT with Stone-Wales defect') pylab.xlabel('Strain (%)') pylab.ylabel('Stress (GPa)') pylab.grid(True) pylab.savefig("mdtrajectory_cnt_fig.png")
现在可以执行脚本。你可以下载以下脚本来确保你做了正确的编辑:
mdtrajectory_cnt.py
点击![]() 按钮将脚本送到Job Manager
按钮将脚本送到Job Manager![]() ,保存并运行。这个任务将会在笔记本电脑上花费3分钟左右。
,保存并运行。这个任务将会在笔记本电脑上花费3分钟左右。
可视化和结果分析
在MD模拟运行中的纳米管长度和应力以不同的ID (“C length”, “C stress”, 和“C stresses factored”)被保存在 .nc文件中。这些将被用于产生名为mdtrajectory_cnt_fig.png的图,这个图将会被自动保存在项目文件夹里。它显示了纳米管的应力应变关系,如下所示。对小应变的线性(弹性)行为使得纳米管杨氏模量(定义为E = stress/strain)的计算成为可能。
通过这个程序,具有一个Stone–Wales缺陷的碳纳米管的杨氏模量计算值大约为2.3TPa(对不同的MD模拟会有变化)。这与无缺陷纳米管杨氏模量计算值1.28TPa[SPAS+99]差得很远。然而,正如[KDE+98]所示,有缺陷的纳米管总的来说具有很高的杨氏模量。
注意!
上图显示对于小的应变,应力是负值,这意味着直到1.1%的应变之前纳米管实际上受压应力。 在MD模拟之前运行几何优化(geometry optimization)可以避免这个问题,可以通过Script Generator中的Optimization模块来完成。然而,对于本实例研究的应变,CNT显示了一个线性的应力-应变行为,所以先运行几何优化所得到的杨氏模量不会有显著的改变。
数据文件mdtrajectory_cnt.nc 会在LabFloor中显示。选中这个文件并点击右手边插件面板上的Movie Tool。Movie Tool可以被用来查看MD轨迹;它将显示一个模拟动画和能量以及温度的数据。如下图所示。
提示!
在动画上右击鼠标点击Show bonds可以将碳原子之间的成键可视化。
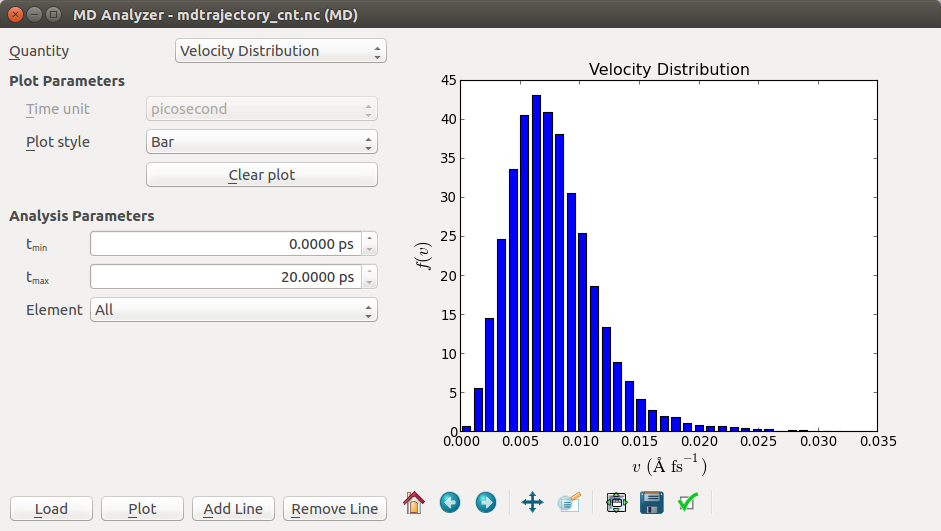
另一个有用的插件为MD Analyzer,它可以显示系统中原子的径向分布,速度分布和动能分布。速度分布的一个屏幕截图如下所示。
参考文献
[KDE+98] (1, 2) A. Krishnan, E. Dujardin, T. W. Ebbesen, P. N. Yianilos, and M. M. J. Treacy. Young’s modulus of single-walled nanotubes. Phys. Rev. B, 58:14013–14019, Nov 1998. doi:10.1103/PhysRevB.58.14013.
[SPAS+99] Daniel Sánchez-Portal, Emilio Artacho, José M. Soler, Angel Rubio, and Pablo Ordejón. Ab initio structural, elastic, and vibrational properties of carbon nanotubes. Phys. Rev. B, 59:12678–12688, May 1999. doi:10.1103/PhysRevB.59.12678.
本文翻译:王吉章