目录
动力学蒙特卡洛kMC模拟
在本教程中,我们将使用基于 AMS 软件图形窗口(GUI),介绍使用 Zacros 程序进行动力学蒙特卡罗(KMC)计算。GUI 将生成一个 pyZacros 脚本(详细参考 pyZacros 手册),该脚本可以像其他运行脚本一样通过 AMSjobs 运行。如果有一些 python 经验,将此脚本扩展到更高级的任务应该是没有困难的。
使用Zacors发表文章,需引用如下:
- Stamatakis, M. and D. G. Vlachos (2011). “A Graph-Theoretical Kinetic Monte Carlo Framework for on-Lattice Chemical Kinetics.” The Journal of Chemical Physics, 134(21): 214115.
- Nielsen, J., M. d’Avezac, J. Hetherington and M. Stamatakis (2013). “Parallel Kinetic Monte Carlo Simulation Framework Incorporating Accurate Models of Adsorbate Lateral Interactions.” The Journal of Chemical Physics, 139(22): 224706.
- Ravipati, S., Nielsen, J., d’Avezac, M., Hetherington, J. and M. Stamatakis (2020). “A Caching Scheme to Accelerate Kinetic Monte Carlo Simulations of Catalytic Reactions”. The Journal of Physical Chemistry A, 124(35): 7140-7154. [please cite if you are using the caching scheme]
- Savva, G. D. and M. Stamatakis (2020). “Comparison of Queueing Data-Structures for Kinetic Monte Carlo Simulations of Heterogeneous Catalysts”. The Journal of Physical Chemistry A, 124(38): 7843-7856. [please cite if you are using the skip-list or the pairing heap queueing system]
- Ravipati, S., Savva, G. D., Christidi, I.-A., Guichard, R., Nielsen, J., Réocreux, R., and Stamatakis, M. (2022). “Coupling the Time-Warp algorithm with the Graph-Theoretical Kinetic Monte Carlo framework for distributed simulations of heterogeneous catalysts”. Computer Physics Communications, 270: 108148 [please cite if you are using the MPI Time- Warp algorithm implementation]
本教程中,我们将使用 Ziff-Gulari-Barshad 模型(该模型的介绍参考资料1、2)。
本教程产生的作业文件:下载并解压得到 *.kin 文件(注意参数与教程中略有差别,不同之处建议以教程为准),在 Zacros 窗口 File → Open 可以打开该文件(注意路径中不要包含中文、空格)
用户需要安装 Zacros-post 包:联网状态下,SCM → Package → 选中 Zacros post 点击 Install 安装。
第一步:一般性参数设置
打开软件图形窗口:AMSJobs → SCM → Kinetics → 点击MKMcxx 改为 Zacros模块(以下称呼为 AMSdynamics 图形窗口)。
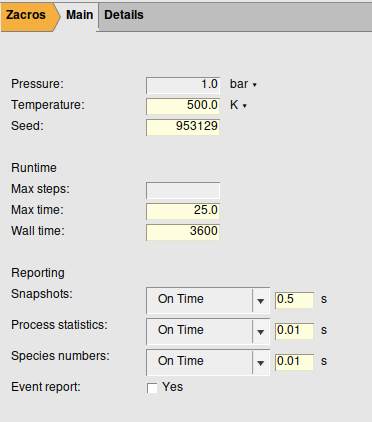
在第一步中,我们将进行常规计算参数设置,位于图形窗口最右侧的面板,一般设置包括系统的条件,如温度和压力,也可以指定随机种子数,如果指定相同随机数,则多次运行也将产生相同的结果。
Runtime 部分的参数,是指定停工条件,可以设定模拟步数或、计算时间,或实际的“墙钟”时间长度,如果不设置则使用默认最大值。
Reporting 是设置采样操作的条件,包括快照操作、过程统计操作和物种数量报告操作,一般按 On Event 对测试运行是最有用的,或按 On time(即步长)设置。
GUI中的所有常规设置都相当于 simulation_input.dat 中的 Zacros 输入选项。这些选项将在脚本中作为 pyZacros Settings 对象生成。本例中使用以下设置:
第二步:设定物种
用户可以在最左侧面板上,添加气体(Gas species)和表面物种(Surface species)。首先添加气体物种:使用 + 按钮,可以添加新的气体物种。也可以从预先格式化的 Spreadsheet 数据表格中复制,然后到这个界面粘贴,从而不需要一个一个手动输入数据。字段可以为空,气体能量的默认值为0.0 eV,初始摩尔分数为0.0。
首先设置气体物种 Gas species,例如:
其中:
- 摩尔比是指初始摩尔比
- 留白的数据,将使用默认值 0.0,例如 CO$_2$ 的初始摩尔为0.0,CO、O$_2$ 的初始摩尔比为 0.45:0.55
- Gas energy、Cluster energy、Activation energy是指气体、Cluster相对于参考集的能量,例如本例中CO、O$_2$是参考集中的物质,因此他们的Gas energy为0.0,活化能与 DFT 计算活化能类似,不过只是过渡态与反应物的单点能之差,详细介绍参考:DFT中的能量如何转化为Zacros动力学蒙特卡洛中的输入能量
接下来,添加表面物种Surface species。默认情况下,总是有空的site,标记为 *。表面物种名称的末尾通常有 * 字符,表示物种的“齿”。表面物种命名遵循这个惯例的话,AMSdynamics 图形窗口能正确处理“齿”数。
其中 CO* 即表示形成的 CO 吸附质,O* 表示一个氧原子形成的吸附质。
在后面的步骤中,气体和表面物种将用于建立 Clusters 和 Reactions。在设置 Clusters 和 Reactions 之后,仍然可以修改或添加新 species。在Zacros中,species 是 simulation_input.dat 文件的一部分,并以相同方式处理。它们将在脚本中生成为 pyZacros Species。在pyZacros中,Molar fractions 是 Settings 对象的一部分,用于更高级的脚本。
第三步:设定晶格结构
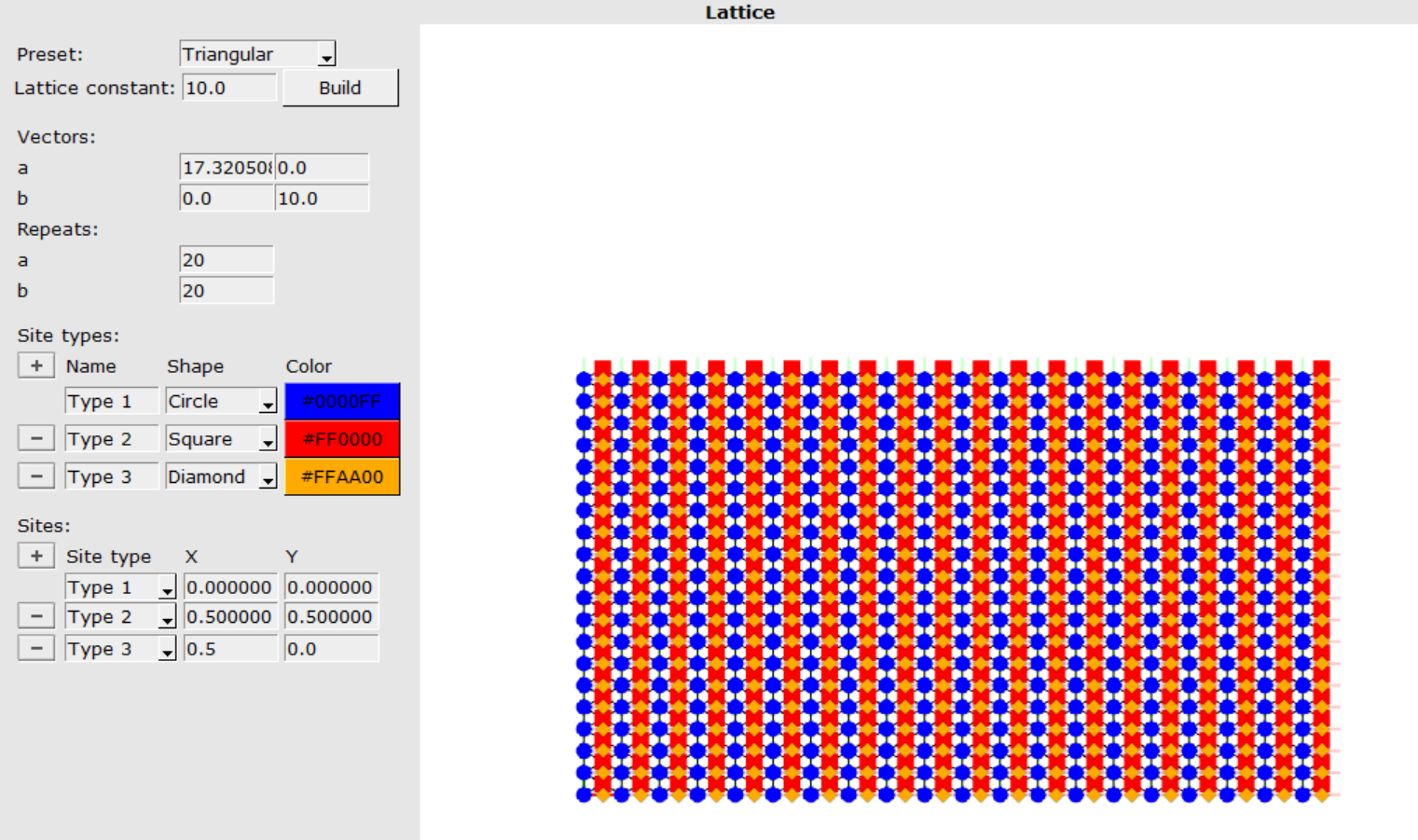
晶格结构位于窗口中间面板,左边是构建晶格的工具。可以使用“Preset”模板列表中的晶系,指定晶格常数后,然后单击“构建”以创建新晶格。这个操作将删除之前创建好的晶格、Clusters 和 Reactions,因为Clusters 和 Reactions 往往和被删除晶格是关联起来的。用户也可以自己创建 Preset:创建晶格(设定好晶格矢量、超胞倍数)后 File → Preset 可以将其(整个超胞)保存为 Preset(下次使用这个 Preset 时,它是作为一个大型单胞的角色了),也可以在 File → Preset → Delete 删除Preset。
晶格矢量可以具有自定义值,也可以根据需要,沿用 Preset 自带值。Repeats 可以在任何时候进行修改,所以使用 Preset 时没有必要去设定。如果使用的是完整的自定义晶格,则可以将其保持为1。
最后是关于位点 Site 的设置,Site types 和Sites 分别设定位点的标记符号,以及位点的相对于单胞的分数坐标。例如:
第四步:指定能量学模型 — Cluster
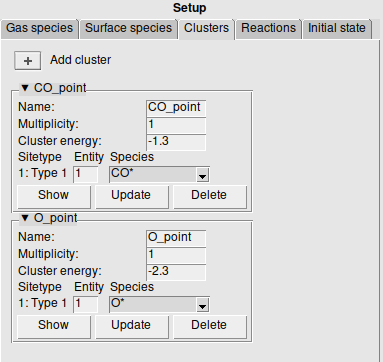
Clusters 设置位于左侧面板的 Clusters 选项卡下面,以“图”的形式表示,可以通过从晶格中选择(鼠标左键可以多选,取消选择时只需在空白处点击即回到没有选择的状态)一个或多个相连接的位点来定义,这样可以确保“图”实际存在于晶格中,并立即提供“图”的可视化效果。选中需要的位点后,单击 + 添加新 Cluster,例如:
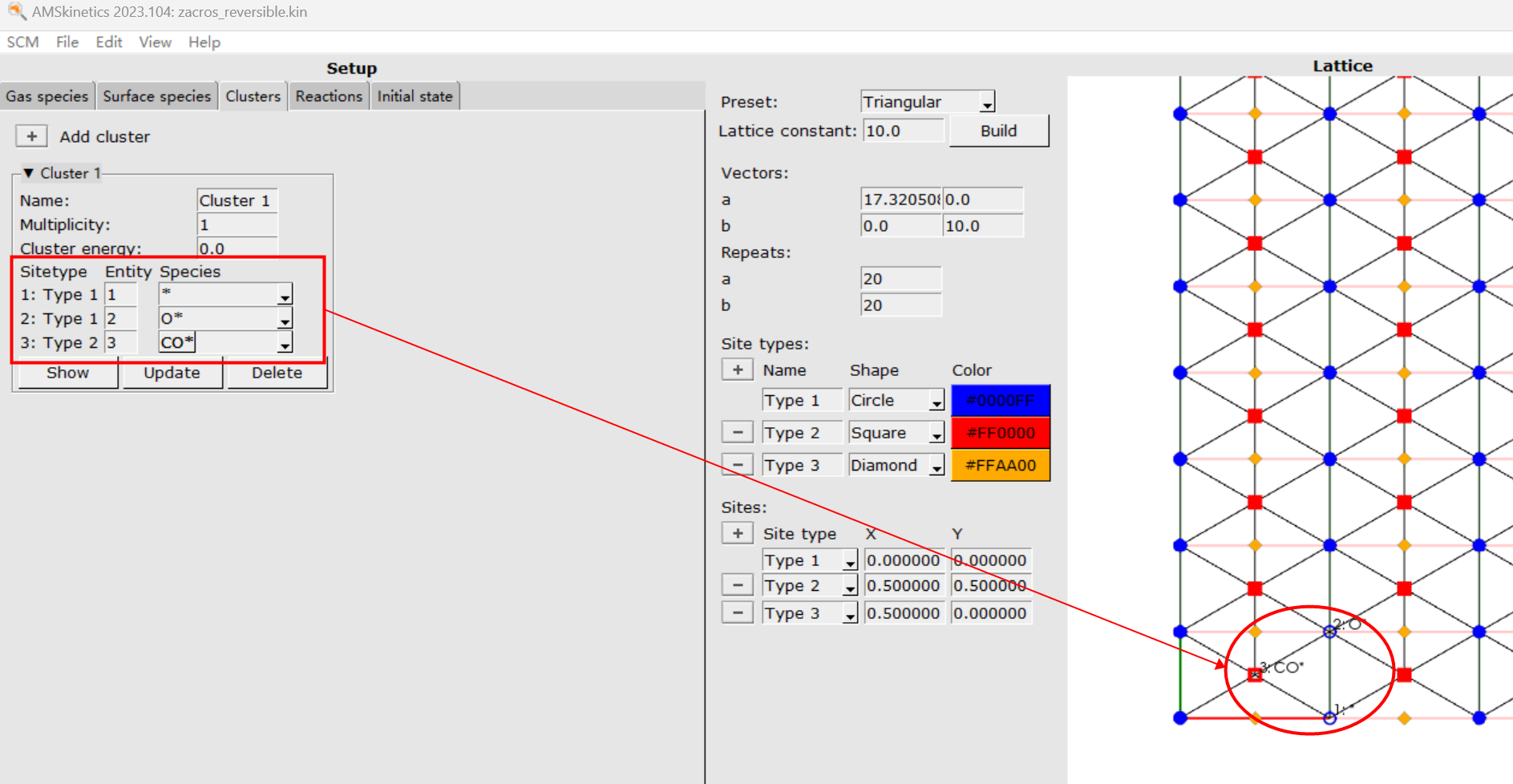
- Name:为该 Cluster 命名,建议避开中文字符。
- Multiplicity:在晶格上找到该 Cluster 的次数。
- Cluster energy:Cluster 的能量。如果有多个 Cluster,即 Multiplicity>1,则每个 Cluster 的 Cluster energy 为 Multiplicity 分之一。如果 Cluster energy 已经归一化,则 Multiplicity 应设为1。
- Sitetype 列出所有 site 的类型,Entry 只是一个从1开始的编号,Species 则为该位点吸附的物种,用户可以自行选择,其中多齿物种在每个结合位点上应该具有相同的 Entity 编号,最终保存设置时,GUI 将检查是否一致。
- 点击 Show 按钮,可以在晶格上显示该 Cluster 在晶格上的位置和标记,一般会在最靠近原点的位点显示 Cluster 代表。
- 可以重新选择新的位点,然后点击 Update 按钮,将 Cluster 的图更改为新选择。
- Delete 按钮删除该 Cluster。
这里我们添加两个 Cluster:CO* 和 O*
第五步:指定反应机制 — Reactions
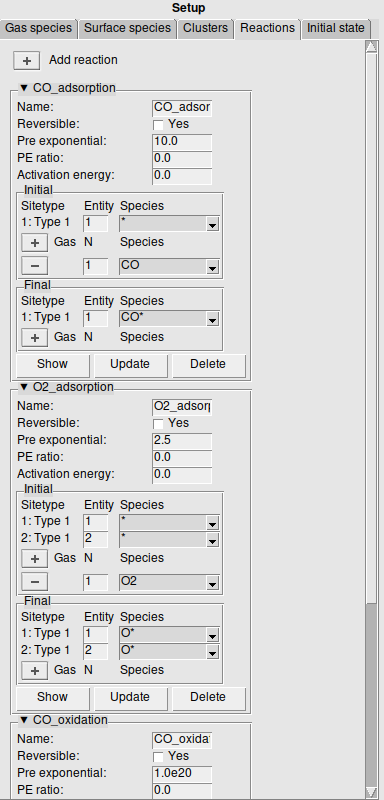
在左侧面板的 Reactions 选项卡下设置,就像 Cluster 一样,反应以“图”表示,并通过相同的方法进行定义。每个位点都代表一个基本反应事件的初始态和终态(详见下图中的具体设置,有助于理解这句话)。
- 勾选 Reversible 复选框,则表示该反应是可逆的(如DFT中的能量如何转化为Zacros动力学蒙特卡洛中的输入能量所述,Zacors 能够自动处理逆反应的活化能)。
- Pre exponential:正向反应 Arrhenius 公式的指前因子。
- PE ratio:对于可逆反应,指定正向和反向指前因子的比率。
- Activation energy:零覆盖极限下的活化能(活化能的含义如前所述,详见DFT中的能量如何转化为Zacros动力学蒙特卡洛中的输入能量)。
然后分别对初态和末态指定吸附物种、可能的气体物种。与 Cluster 一样,多齿物种的 Entity 编号应该相同。点击 Gas 前面的 +,即可增加的气体物种,N 列为气体物种的编号列。Show、Update 和 Delete 的工作方式与 Cluster 相同。
我们将添加 3 个反应;两个吸附反应和一个氧化反应:
在 Zacros 中,反应机制设置位于 mechanism_input.dat 文件中,将生成 pyZacros ElementaryReaction。
第六步:运行并分析结果
本教程没有使用左侧面板上的 Initial state 选项卡,它可以用于设定随机或指定初始状态的表面物种填充位点。现在可以 File → Save 保存作业,将生成 *.kin 文件,该文件存储 GUI 中的各种设置,稍后可以由 AMSdynamics 的图形窗口再次打开类似于 AMSinput 的 *.ams 文件。同时将生成运行脚本 *.run 文件,其中包含完整的 pyZacros 脚本。
File → Run 运行作业。
pyZacros 生成的所有文件和 Zacros 的输出都位于 *.results 目录中。如果 AMSdynamics 窗口如果仍然是打开的状态,它会弹出提示,要求分析结果。当然也可以通过菜单栏 View → Results 来查看结果。从而打开 Zacros post 窗口。在 Zacros post 中,您可以选择 Plot → Species Numbers 显示物种随时间变化的曲线:
其它分析,用户可以自行探索。
参考资料
- 本文参考官方英文教程:Kinetic Monte Carlo