adf:geoopt2019
目录
如何优化分子的几何结构
软件版本为AMS2019.3及之前的版本,AMS2020以后的版本请参考链接:优化分子的几何结构
此处以氧气分子举例:优化中性<chem>O2</chem>分子:
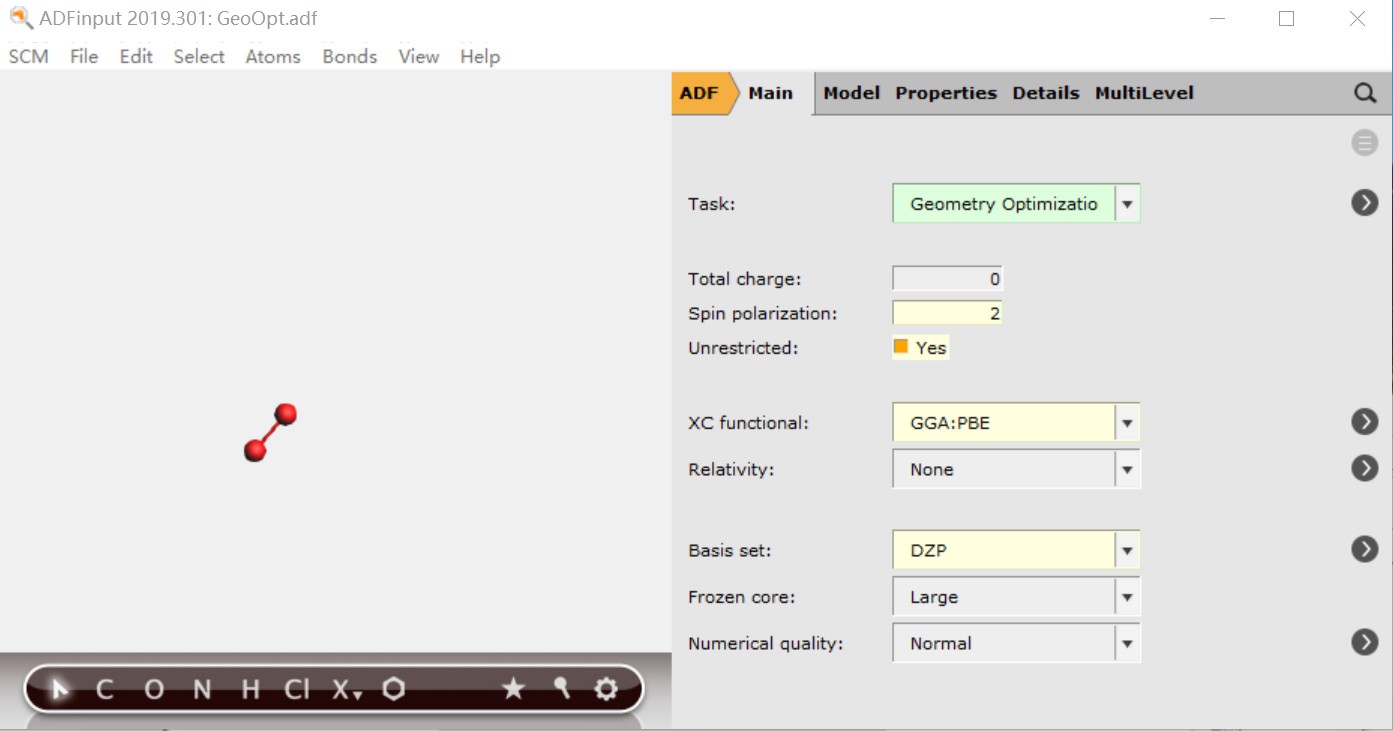
参数设置
注意
- O2的基态是三重态(两个电子未配对),因此Spin Polarization设为2(其他例如水分子,基态是单重态,所有电子配对,那么Spin Polarization则为默认值0)
- 体系基态处于单重态还是三重态,一般而言是由二者能量决定的,谁的能量低,就是谁;
- 结构优化,除了存在范德华力之外,一般对泛函、基组不太敏感,不同的参数优化出来结果几乎差别很小,而这样小的差别往往对其他性质的计算没有影响;
- 大体系的优化,为了提高效率,一般并不会一开始就使用精度较高的方法、基组,开始使用低精度的方法、基组,优化结束后,使用收敛的结果,增大基组,使用更高精度的泛函,接着优化,这样能在保证精度的前提下,极大地节省计算时间;
- 结构优化Numerical Quality设置为Normal即可,与Good几乎没有任何差别,但后者计算量比前者大好几倍;
在ADFinput分别点击file-save,file-run,即运行计算。或者提交到服务器计算,具体参考:提交作业
结果查看
检查优化是否成功?
计算结束后,查看结果:SCM - Logfile,尾部:
<Dec04-2019> <22:08:51> current energy -0.35356828 Hartree <Dec04-2019> <22:08:51> energy change -0.00000243 0.00100000 T <Dec04-2019> <22:08:51> constrained gradient max 0.00016137 0.00100000 T <Dec04-2019> <22:08:51> constrained gradient rms 0.00009317 0.00066667 T <Dec04-2019> <22:08:51> gradient max 0.00016137 <Dec04-2019> <22:08:51> gradient rms 0.00009317 <Dec04-2019> <22:08:51> cart. step max 0.00003214 0.01000000 T <Dec04-2019> <22:08:51> cart. step rms 0.00001856 0.00666667 T <Dec04-2019> <22:08:51> GEOMETRY CONVERGED <Dec04-2019> <22:08:51> Calculating Energy Terms for Final Geometry Coordinates in Geometry Cycle 8 Atom X Y Z (Angstrom) 1.O 0.000000 0.000000 -0.616495 2.O 0.000000 0.000000 0.616495 <Dec04-2019> <22:08:51> >>>> CORORT <Dec04-2019> <22:08:51> >>>> CLSMAT <Dec04-2019> <22:08:51> >>>> ORTHON <Dec04-2019> <22:08:51> >>>> GENPT <Dec04-2019> <22:08:51> Block Length= 127 <Dec04-2019> <22:08:51> >>>> PTBAS <Dec04-2019> <22:08:51> >>>> CYCLE <Dec04-2019> <22:08:51> |Error| MaxErr Wt(A-DIIS) <Dec04-2019> <22:08:51> 1 0.00000001 0.00000000 <Dec04-2019> <22:08:51> 2 0.00000000 0.00000000 100.0 <Dec04-2019> <22:08:51> SCF converged <Dec04-2019> <22:08:51> 3 0.00000000 0.00000000 100.0 <Dec04-2019> <22:08:51> >>>> TOTEN <Dec04-2019> <22:08:52> >>>> POPAN <Dec04-2019> <22:08:52> >>>> DEBYE <Dec04-2019> <22:08:52> >>>> AMETS <Dec04-2019> <22:08:52> >>>> POPUL <Dec04-2019> <22:08:52> Bond Energy -0.35356828 a.u. <Dec04-2019> <22:08:52> Bond Energy -9.62108233 eV <Dec04-2019> <22:08:52> Bond Energy -221.87 kcal/mol <Dec04-2019> <22:08:52> NORMAL TERMINATION <Dec04-2019> <22:08:52> END
注意:
- 优化过程会不断地计算不同的分子结构,直到找到最低能量的结构为止。
- energy change表示优化过程中,能量的变化量
- constrained gradient max表示最大能量梯度
- constrained gradient rms表示能量梯度均值
- cart. step max表示原子的最大位移量
- cart. step rms表示原子平均位移量
- 上述五个标准都达标了后面就会有5个T,F表示未达标,字母前面的数值是判断标准
- 一般而言,第二个标准最重要,也往往是最后一个达标的。默认标准为0.001,已经是一个比较严格的标准了
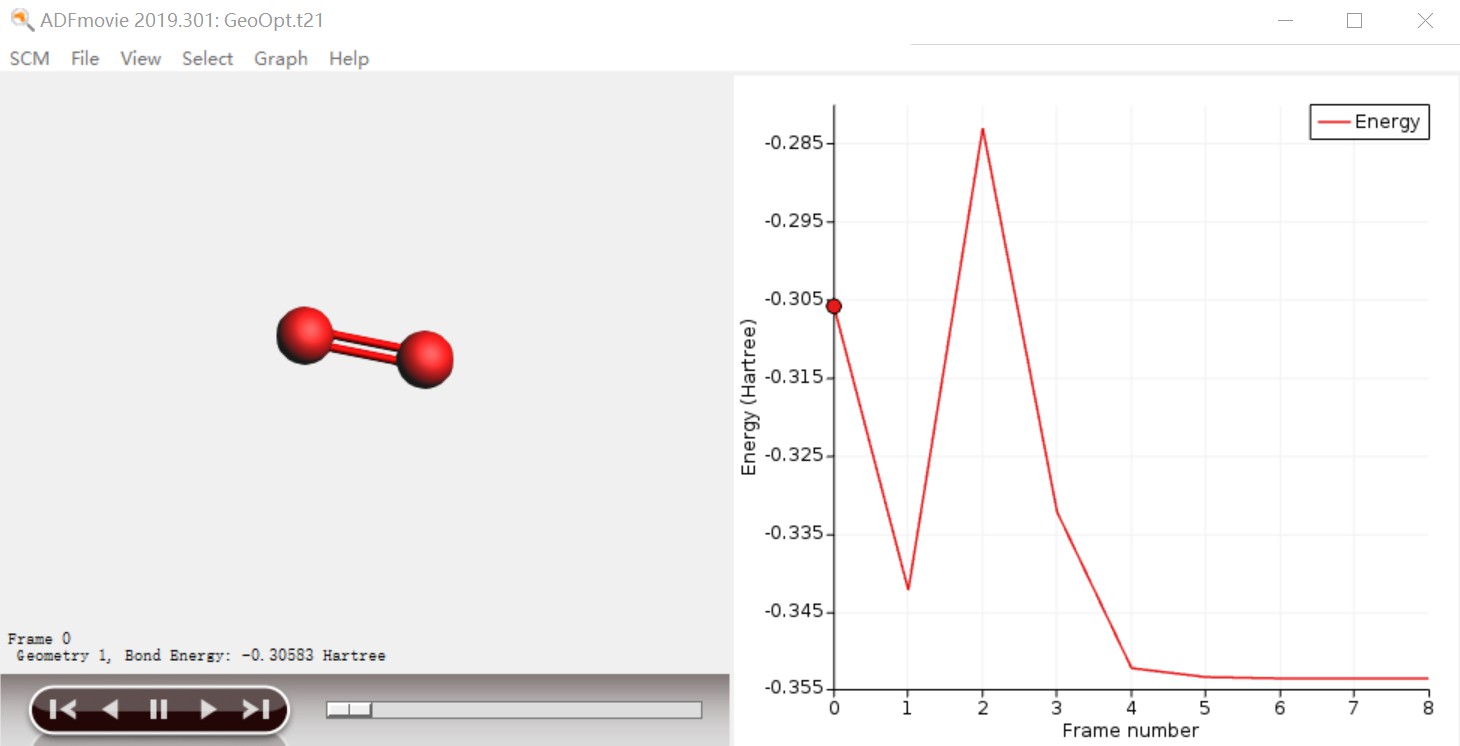
查看优化过程结构、能量变化
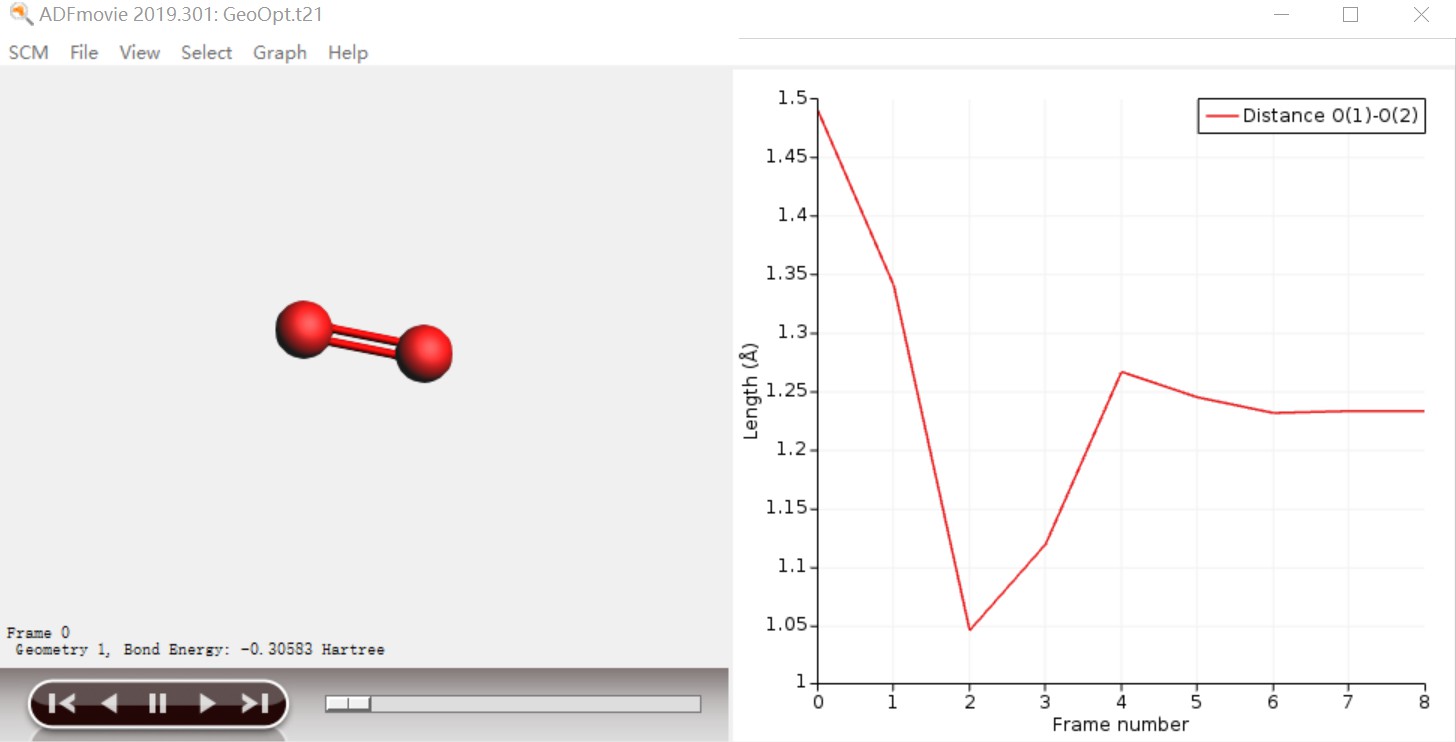
查看键长键角、二面角的变化
Graph - delete graph,删掉之前的能量变化曲线。按住shift键,即可选择多个原子,如图所示,安住shift键,分别选中两个O原子,原子变为高亮,表示被选中。然后Graph - Distance, Angle, Dihedral,即可查看键长的变化曲线,如果选中三个、四个原子,则显示对应的键角、二面角的变化曲线:
右方出现键长变化的图,窗口下方也会出现每一步的具体键长数值。
注意:键角、二面角也是类似,选中对应的原子,然后选择上面的选项即可。
如果要将某一帧(往往需要导出最后一帧)的结构导出,参考:
到此优化过程完成!
一般结构优化完成之后,会进行频率计算,检查是不是存在虚频。如果存在虚频,则表示优化不精确,需要继续优化。但这个问题在ADF一般很少出现。因此,对于非弱键的体系,都可以省去这一步。如果不放心,可以进行频率计算。
AMS软件提供免费试用,试用申请方式参见:AMS免费试用
adf/geoopt2019.txt · 最后更改: 2022/10/31 19:20 由 liu.jun