目录
使用Metadynamics动力学方法研究 Cu(111) 中 Cu 空位的扩散(PLUMED)
版本:P-2019.03
本教程介绍如何使用 PlumedMetadynamics 类和 PLUMED 插件在 QuantumATK 中设置和运行 Metadynamics 模拟,介绍如何研究 Cu(111) 表面上 Cu 空位扩散相关的自由能分布,以及如何设置 Metadynamics 计算以及如何分析结果。
介绍
Metadynamics [LP02] 是基于分子动力学的一种很有用的模拟方法,可以研究多维自由能表面(FESs),通过基于体系微观坐标的多个综合变量(Collective Variables,CV)的函数重构体系的 FES。
在 Metadynamics 模拟过程中,会在模拟期间定期向体系中添加附加的“歧视”势能(bias potential)。这可以使体系能够克服很高势垒,逃离较深的自由能极小值,从而有效地探索整个自由能表面。在材料科学领域,该方法已用于研究晶体多态性、固液界面以及固体和表面的化学反应。本教程将应用这种方法研究 Cu(111) 表面上 Cu 空位的扩散。
QuantumATK 通过 PLUMED 插件实现 Metadynamics。在本教程中,我们先简要地介绍该方法,然后演示如何为 Cu(111) 上的空位扩散设置 Metadynamics 脚本,以及如何分析生成的自由能表面。
关于 Metadynamics 方法的更多信息,可参见参考文献 [BBP11] 和 [BN15]。
理论背景
在 Metadynamics 中,在特定的自由度相空间中构造外加的歧视势能,$S$,这些特定的自由度通常称为综合变量(CVs)[LP02]。$S$ 是一系列 d 个体系微观坐标 $R$ 的函数。
$$S(R) = (S_1 (R), ..., S_d (R)).$$
势能是在时间 t 内,高斯函数沿 CV 空间中轨迹的积分。在文献 [BBP11] 中,简单的势能公式为:
$$V(S,t) = \int_{0}^{t} dt^{'} \frac{W}{\tau} \exp\left( -\sum_{i=1}^{d} \frac{(S_i (R)-S_i({R}(t^{'} )))^2}{2\sigma_i^2} \right),$$
这里,$\tau$ 是高斯函数积分的步幅,$\sigma_i$ 是在第 $i$ 个 CV中高斯函数的宽度,$W$ 是高斯函数的高度。歧视势能的作用是使系统远离当前的局部极小值,并使其能够到达相空间的新区域。
Cu(111) 中 Cu 空位的 Metadynamics 动力学模拟
在 Cu(111) 上创建一个空位
想要在 Cu(111) 上构造一个 Cu 空位,首先您需要创建一个 Cu(111) 面。
在 ![]() Builder,点击 Add
Builder,点击 Add ![]() From Database,导入“Copper”。然后点击 Builders
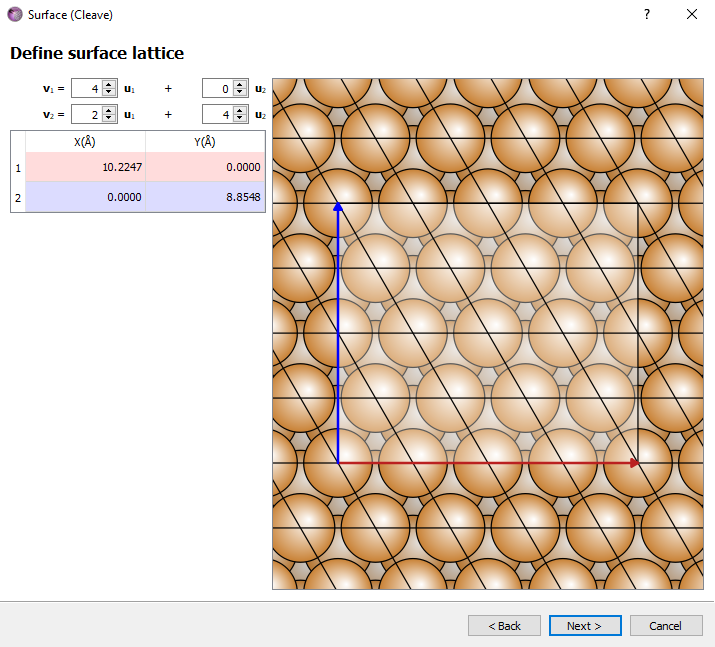
From Database,导入“Copper”。然后点击 Builders ![]() Surface (Cleave),利用以下设置构造一个 Cu(111) 面:
Surface (Cleave),利用以下设置构造一个 Cu(111) 面:
- 设置 Miller 指数为 <111>
- 采用下图所示的表面晶格:
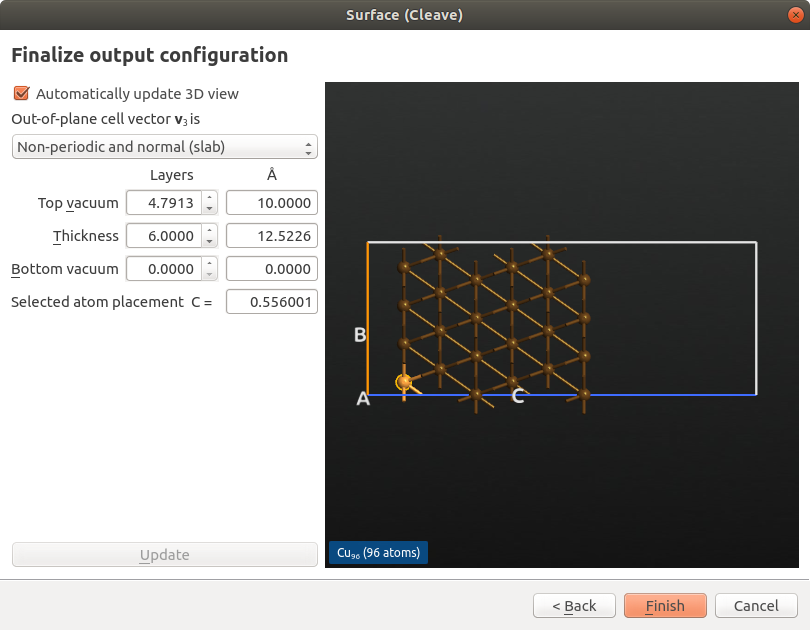
- 设置平面外晶胞矢量为“Non-periodic and normal (slab)”
- 设置厚度为 6 层,真空间隙为 10 Å。
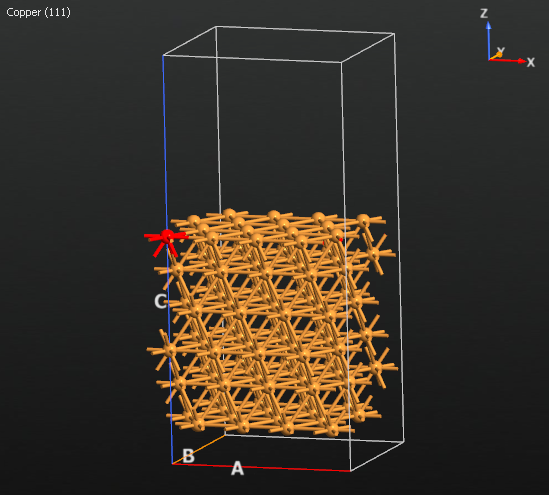
接下来,删除最上面那层坐标为 $(x,y) = (0,0)$ 的原子(下图中红色部分),创造出一个空位:
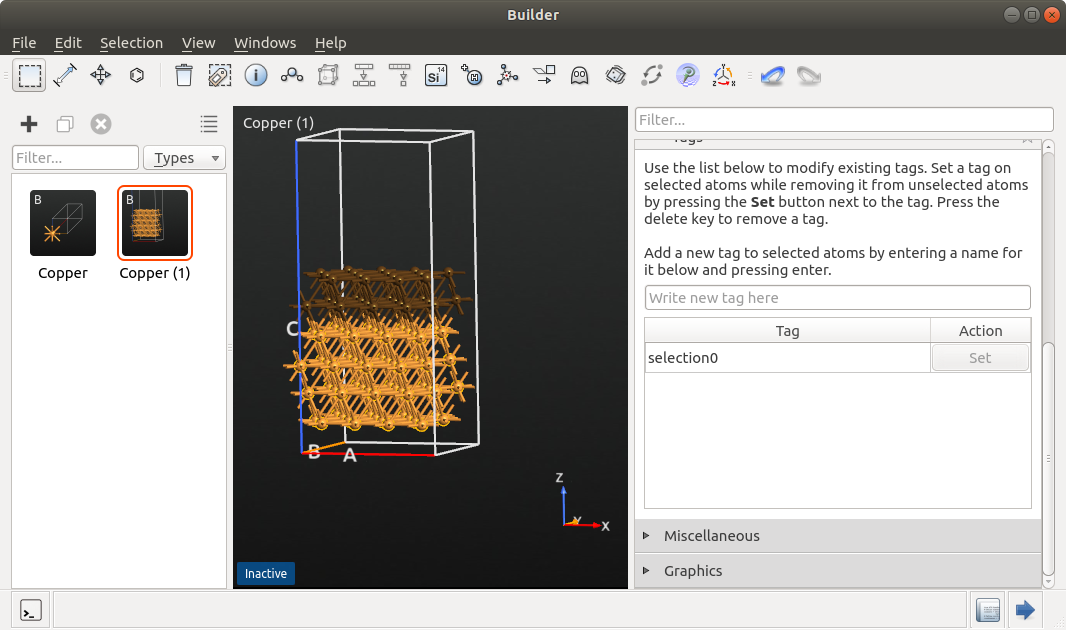
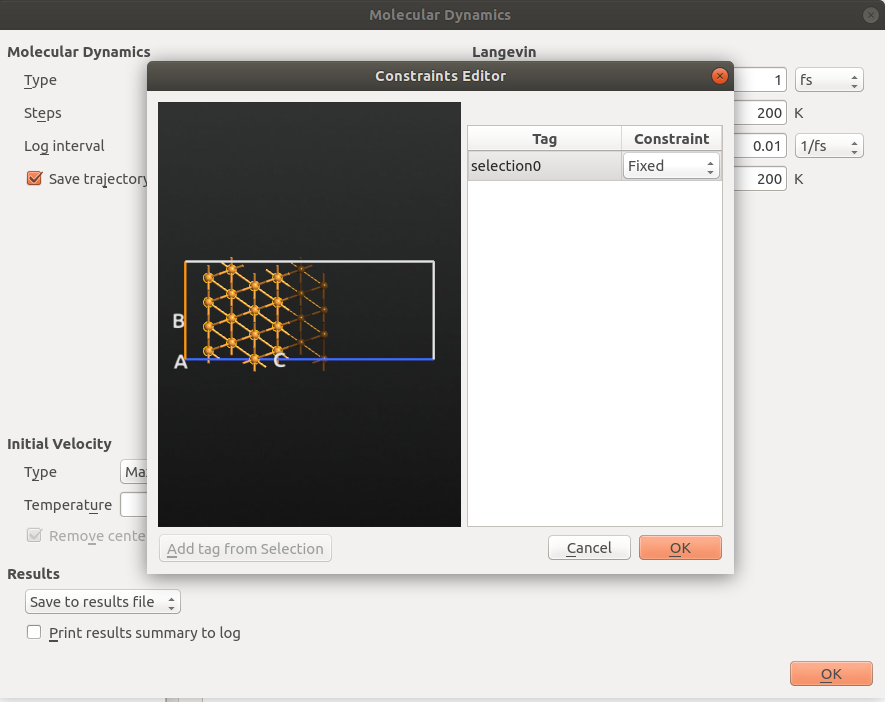
最后,点击 Selection Tools ![]() Tags,标记 Cu(111) 最下面的 4 层。此标记将用于在 metadynamics 模拟过程中对它们的固定。
Tags,标记 Cu(111) 最下面的 4 层。此标记将用于在 metadynamics 模拟过程中对它们的固定。
创建 Metadynamics 脚本
现在您可以开始创建 metadynamics 模拟的脚本了。将结构从 Stash 发送到 ![]() Script Generator ,按照如下设置脚本:
Script Generator ,按照如下设置脚本:
1.添加以下模块
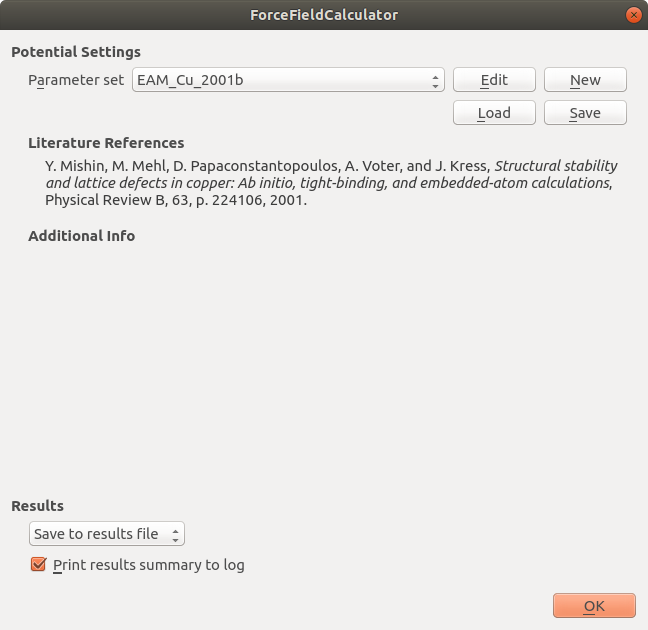
2.双击 ![]() ForceFieldCalculator 模块,使用“EAM_Cu_2001b”参数组。
ForceFieldCalculator 模块,使用“EAM_Cu_2001b”参数组。
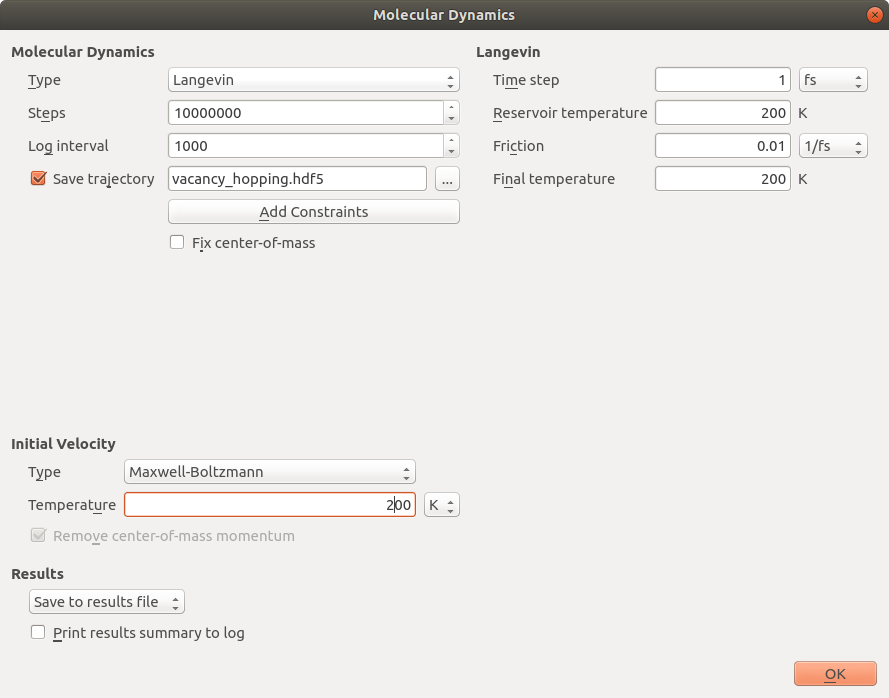
3.打开 ![]() MolecularDynamics 模块,如下图设置模拟参数:
MolecularDynamics 模块,如下图设置模拟参数:
4.最后,点击 Add Constraints,固定 Cu(111) 最下面已被标记的那 4 层。
点击 ![]() Scripter 里的
Scripter 里的 ![]() 按钮,将脚本发送到
按钮,将脚本发送到 ![]() Editor。然后添加下文所示高亮部分,包含了执行 metadynamics 计算的
Editor。然后添加下文所示高亮部分,包含了执行 metadynamics 计算的 PlumedMetadynamics 类。
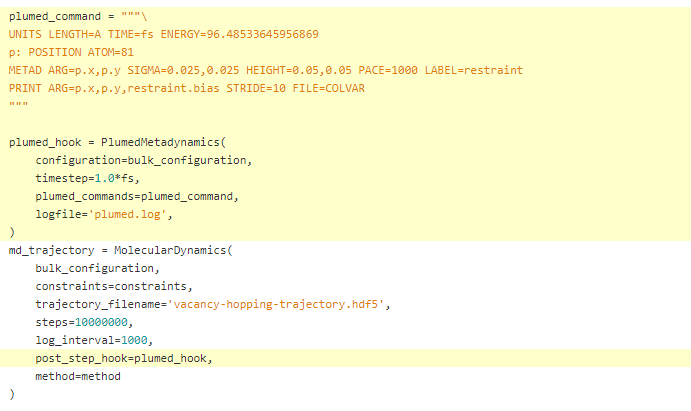
# ------------------------------------------------------------- # Calculator # ------------------------------------------------------------- potentialSet = EAM_Cu_2001b() calculator = TremoloXCalculator(parameters=potentialSet) calculator.setVerletListsDelta(0.25*Angstrom) bulk_configuration.setCalculator(calculator) nlprint(bulk_configuration) bulk_configuration.update() nlsave('vacancy-hopping.hdf5', bulk_configuration) # ------------------------------------------------------------- # Molecular Dynamics # ------------------------------------------------------------- initial_velocity = MaxwellBoltzmannDistribution( temperature=200.0*Kelvin, remove_center_of_mass_momentum=True ) method = Langevin( time_step=1*femtoSecond, reservoir_temperature=200*Kelvin, friction=0.01*femtoSecond**-1, initial_velocity=initial_velocity, heating_rate=0*Kelvin/picoSecond, ) fix_atom_indices_0 = [ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63] constraints = [FixAtomConstraints(fix_atom_indices_0)] plumed_command = """\ UNITS LENGTH=A TIME=fs ENERGY=96.48533645956869 p: POSITION ATOM=81 METAD ARG=p.x,p.y SIGMA=0.025,0.025 HEIGHT=0.05,0.05 PACE=1000 LABEL=restraint PRINT ARG=p.x,p.y,restraint.bias STRIDE=10 FILE=COLVAR """ plumed_hook = PlumedMetadynamics( configuration=bulk_configuration, timestep=1.0*fs, plumed_commands=plumed_command, logfile='plumed.log', ) md_trajectory = MolecularDynamics( bulk_configuration, constraints=constraints, trajectory_filename='vacancy-hopping-trajectory.hdf5', steps=10000000, log_interval=1000, post_step_hook=plumed_hook, method=method ) bulk_configuration = md_trajectory.lastImage() nlsave('vacancy-hopping.hdf5', md_trajectory)
您也可以在此处下载输入文件:↓ vacancy-hopping.py。
在以上输入中,plumed_command 部分包括被直接传递到 PLUMED [BBP11] 的输入。 其中有下面四个命令行:
- UNITS:这行控制 PLUMED 中采用的单位。如上编辑此行,将会用到 Å、fs 和 eV 这些单位。
- p:这行是用来定义约束的原子,在本例中是序号为 $81$ 的原子。
注意
这个数值是根据 PLUMED 中使用的序号设置的,在 QuantumATK 中原子的序号从 $1$ 开始,而不是 $0$。因此这个值应被设置为“序号+1 号原子”,这里的原子序号是指被约束原子的序号。
我们将使用在每个轴上宽度 $0.025$ 、高度 $0.05$ 的高斯函数查看 $x$ 轴和 $y$ 轴上的势能面。
- METAD:这行控制模拟期间 metadynamics 的类型和采用的高斯函数。
- ARG:这个参数用于设置变量集。在本例中,我们将使用的变量集为原子 $p$ 沿笛卡尔坐标 $x\ (p.x)$ 和 $y\ (p.y)$ 方向。
- SIGMA:这个参数用于设置每个变量集所用高斯函数 $\sigma$ 的宽度。本例中,我们为两个变量集都设置为 $\sigma = 0.025$。
- HEIGHT:这个参数用于设置每个变量集所用高斯函数的高度。本例中,我们为两个变量集都设置为 $0.05$ eV。
- PACE:这个参数用于设置添加新高斯函数的间隔。本例我们每 $1000$ MD 步添加一个新的高斯函数。
- LABEL:这个参数用于设置变量集的名称。
- PRINT:这行控制 PLUMED 输出的格式。
- ARG:这个参数控制输出的变量,本例中为 $p.x$、$p.y$ 和偏差。
- STRIDE:表明输出间隔,本例为 10 MD 步。
- FILE:此行指明输出文件名称。
如果您想了解更多详尽信息,我们推荐您查阅 PLUMED documentation。这些命令都被转至 PlumedMetadynamics 类,后面的使用 hook 函数传至 MolecularDynamics 类。
分析结果
除了标准的 QuantumATK 输出,在作业的最后您将获得 COLVAR 和 HILLS 文件。这两个 PLUMED 的输出文件将用于分析 metadynamics 模拟。
为画出自由能 $\mathrm{F}$ 关于综合变量 $\mathrm{CV\ 1}$ 和 $\mathrm{CV\ 2}$ 的函数分布图,首选要用 atkpython extract_F.py 命令运行脚本 ↓ extract_F.py。将会产生这三个输出文件:
F_vs_cv1.dat包含 $\mathrm{F}$ vs. $\mathrm{CV\ 1}$ 的数据。F_vs_cv2.dat包含 $\mathrm{F}$ vs. $\mathrm{CV\ 2}$ 的数据。F_vs_cv1_cv2.dat包含 $\mathrm{F}$ vs. $\mathrm{CV\ 1}$ 和 $\mathrm{F}$ vs. $\mathrm{CV\ 2}$ 的数据。
可以输入以下命令运行脚本 ↓ plot_F_vs_cv1_cv2.py 即可得到自由能分布 $\mathrm{F}$ 的示意图:
atkpython F_vs_cv1_cv2.py
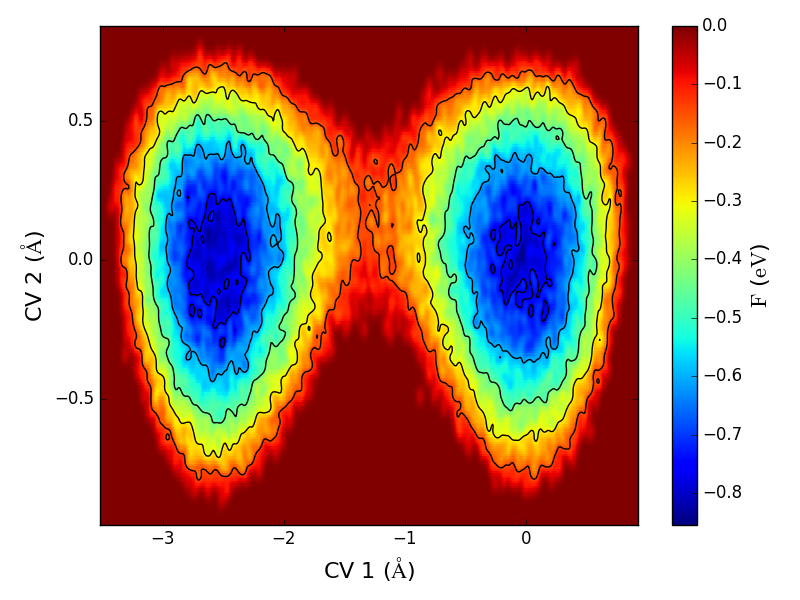
上图显示了自由能分布关于综合变量 $\mathrm{CV\ 1}$ 和 $\mathrm{CV\ 2}$ 的热图。可以看出,在自由能面上有两个极小值,对应于空位从一个位置到相邻位置的扩散。
模拟时间内综合变量的演变可以用以下命令运行脚本 ↓ cv1_cv2_vs_time.py 获得:
atkpython cv1_cv2_vs_time.py
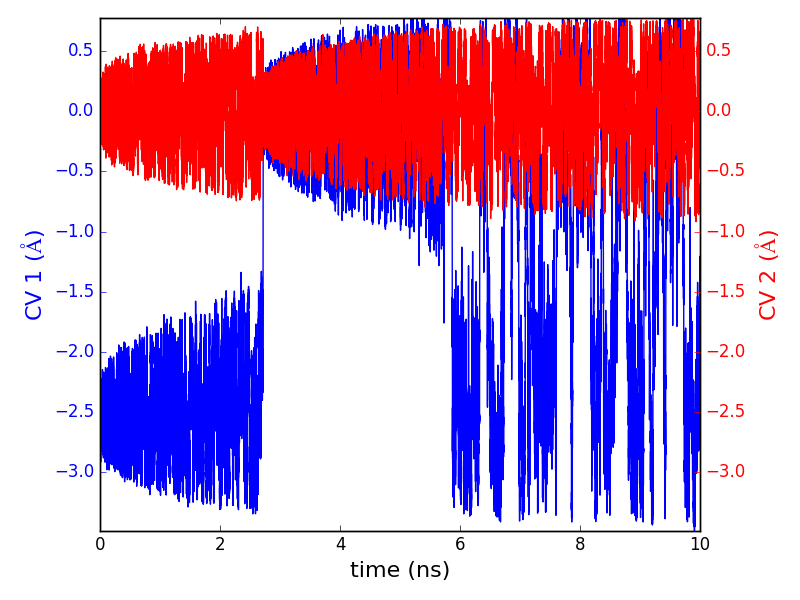
生成图展示了 $\mathrm{CV\ 1}$(红色) 和 $\mathrm{CV\ 2}$(蓝色)关于模拟时间的函数演变。可以看出,$\mathrm{CV\ 2}$ 对应于笛卡尔坐标 $y$,在固定值附近($\mathrm{CV\ 2} = 0 \mathrm{Å}$)振荡,因为两个自由能极小值都出现在相同的 $y$ 值处。相反地,$\mathrm{CV\ 1}$ 对应于笛卡尔坐标 $x$,表明模拟势从左边的极小值($\mathrm{CV\ 1} = -2.5 \mathrm{Å}$)开始。第一个极小值被填充直到接近 $2.2$ ns,然后体系移至第二个极小值($\mathrm{CV\ 1} = 0 \mathrm{Å}$)。第二个极小值也被填充后(大约在 6 ns 内)系统在两个极小值间的振荡概率相同,直到模拟结束。
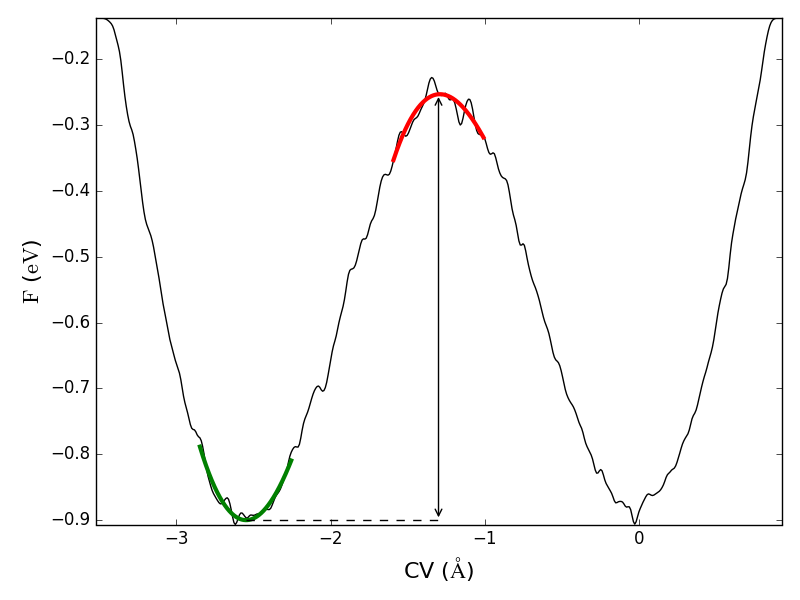
自由能势垒也可以通过运行脚本 ↓ barrier.py 分析:
atkpython barrier.py
结果显示势垒高度为 $0.647$ eV,与 [KNB17] 中结果一致。
参考
- [BN15] Kristof M. Bal and Erik C. Neyts. Merging metadynamics into hyperdynamics: Accelerated molecular simulations reaching time scales from microseconds to seconds. Journal of Chemical Theory and Computation, 11(10):4545–4554, 2015.
- [BBP11] (1, 2, 3) Alessandro Barducci, Massimiliano Bonomi, and Michele Parrinello. Metadynamics. WIREs: Comput. Mol. Sci., 1(5):826–843, 2011. doi:10.1002/wcms.31.
- [KNB17] Suresh Kondati Natarajan and Jörg Behler. Self-diffusion of surface defects at copper-water interfaces. The Journal of Physical Chemistry C, 121(8):4368–4383, 2017.
- [LP02] (1, 2) Alessandro Laio and Michele Parrinello. Escaping free-energy minima. Proc. Natl. Acad. Sci., 99(20):12562–12566, 2002.