文献重现:氢键强度、轨道作用、电荷分析
文献详解:芳香性、杂化情况、静电作用以及共价作用,在Adenine–Thymine碱基对及其类似模仿结构的共振增强氢键中的作用研究
原文:ChemistryOpen 2015, 4, 318 – 327
摘要:本文涉及的ADF计算功能包括
- 二聚体分子几何结构计算;
- 成键轨道组分分析;
- 键能分解;
- 片段分析;
- 片段形成分子过程中电荷流向分析(VDD);
- 查看分子轨道(MO)的形状、空间分布;
- 片段构成分子之后,电子在片段间的转移情况;
- 分子轨道(MO)投射到片段轨道(SFO)上的占据情况;
- 键能分解到各个不可约表示,其中AAA不可约表示实际上是π轨道,AA实际上是σ轨道
- 分析Pauli排斥的大小、轨道相互作用的大小、各个不可约表示中轨道相互作用的大小、色散修正的大小;
- 通过去掉特定对称性的空轨道,来得到特定对称性对能量(轨道相互作用能)、(VDD)电荷的贡献;
- 片段轨道重叠的大小。
引言
氢键在很多生物化学、超分子化学过程中,扮演着重要的角色1)。观察了Rosalind Frankli提供的DNA的X射线衍射图谱,Watson 和Crick在1953年提出:在遗传密码作用机制中,氢键的作用必不可少2)。在DNA中,核苷酸的双螺旋链是由嘌呤与嘧啶之间的氢键作用结合在一起,形成核酸碱基,也就是腺嘌呤-胸腺嘧啶(AT)、鸟嘌呤-胞嘧啶(GC)。
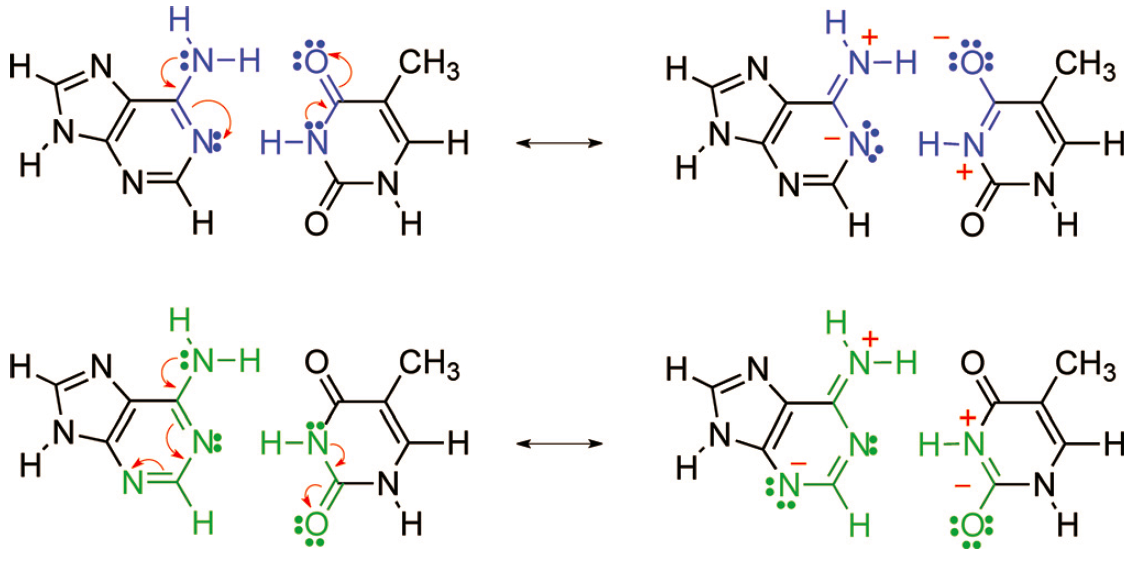
Gilli等人3)认为DNA碱基对中的氢键是由л辅助增强的,也就是所谓的共振辅助氢键(RAHB)。共轭的双键共振,通过电荷离域辅助氢键,使得氢键中施主与受主之间的距离缩短。他们认为RAHB在分子内、分子间均可能发生。关于分子内、分子间的RAHB有大量的理论研究工作4),5)。Gilli等人提出的DNA碱基对的共振辅助示意图如图1(路易斯结构式):
图1 AT中的共振辅助氢键
在之前的发表的文献中6),我们从理论上阐明:静电相互作用和轨道相互作用对DNA碱基对的氢键同等重要;л电子对稳定性提供补充作用。这项结论得到其他人的再次确认7)8)。л电子体系的离域与σ电子体系施主-受主相互作用的协同作用很小。也就是说,л、σ相互作用同时发生,仅仅比各自单独发生的强度之和微微大一点。最近我们发现G-四连体中,染色体端粒部位出现的这种非常有趣的协同行为,是由于σ电子体系施主-受主轨道相互作用导致电荷重新分布所致,而不是由于л电子体系的共振增强9)。其中л离域对氢键只是提供了一个补充性的稳定化作用。
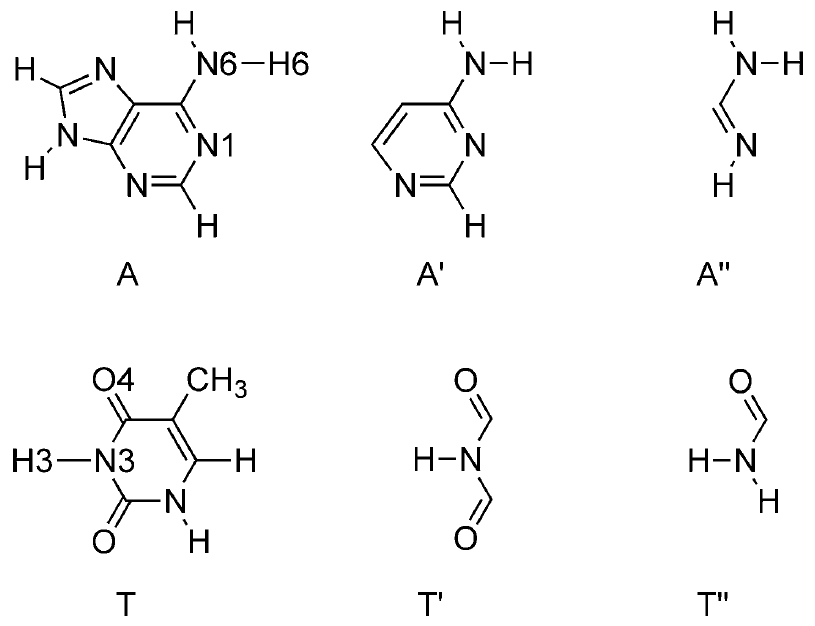
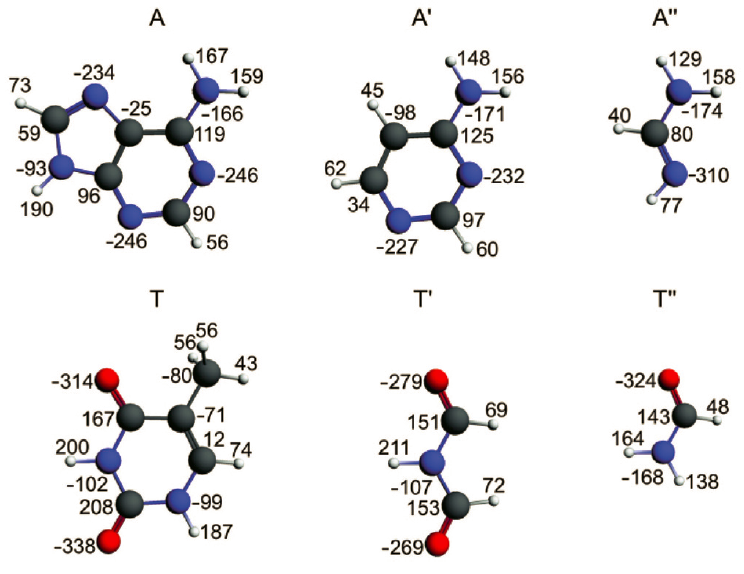
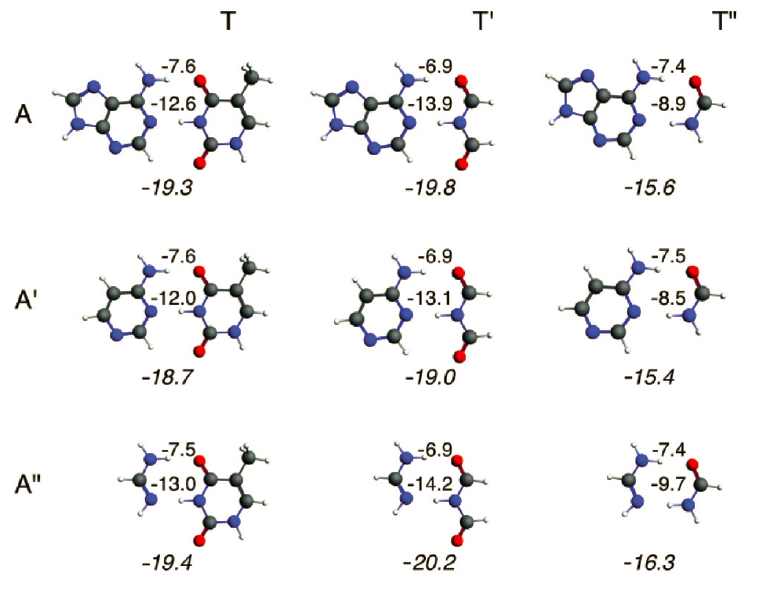
图2 腺嘌呤(A)及其类似物、胸腺嘧啶(T)及其类似物
本文我们研究共振对AT以及更小的类似物(图2)的氢键的辅助作用。因为Gilli的建议认为较小的模仿结构也能得到同样的共振辅助。我们之前对Watson–Crick碱基对进行过密度泛函(DFT)计算,也表明氢键主要影响图1中蓝色部分的原子电荷。然而σ电子的共振却环绕在更大的区域(图1下方的绿色部分),这表明我们可以去掉嘌呤的五元环,但不能去掉六元环;而对于嘧啶,我们需要考虑所有的前线原子。
为了检验共振结构的电荷重排情况,л电子的数量从A到A’、A’’,T到T’、T’’逐渐减少。所有可能的组合(AT、AT’、AT“、A’T、A’T’、A’T”、A“T、A”T’、A“T”)都考虑在内。这个计算研究可以探明л辅助是否只来源于芳香性10),11),或者质子施主、受主原子的sp2杂化已经带来了л电荷离域?从逻辑上顺理成章地,可以通过对比二聚体sp2杂化和sp3杂化的差别,弄清楚杂化的重要性。后一种情况的氢键已知较长——如果差别仅仅是分子的饱和程度而参与形成氢键的前线原子相同(后面章节的比较重能看到)。
对DNA碱基对AT及其更小的模仿结构的计算分析,我们采用的是基于散修正的密度泛函理论(DFT-D3)12)。计算得到的AT及模仿结构在几何结构和成键的微小差别,可以通过的DFT分子轨道以及对应的能量分解(EDA)进行定量分析,从而得到解释。这些结果揭示:л辅助与单体的л电子数目无关,但质子的施主和受主原子拥有л电子是必须的。
计算方法
基本流程
所有计算都是使用ADF2013完成13),14)。分子轨道使用Slater基组TZ2P展开(不含任何Gaussian函数)15)。对于C、N、O原子,冻结了1s轨道16),也就是对应Frozen core:Small。而在自洽场(SCF)计算过程中,计算库伦、交换相关势的时候,软件自动采用一套包含s、p、d、f、g的Slater函数来拟合分子的电子密度17),18)。
泛函采用的是BLYP-D3(BJ),BLYP为GGA泛函的一种,-D3(BJ)是一种对泛函的长程行为进行“经验参数式”修正的所谓“色散修正”方法19)。色散修正主要用于弱相互作用体系,例如氢键、超分子。需要进行色散修正的原因:一般的密度泛函势函数随着离原子核距离增大衰减过快,因此对距离较远的原子的相互作用描述的不好。-D3(BJ)修正之后,原子间长程相互作用势的衰减形式为C6/(R6+c)20)。其中c、C6为拟合得到的常数,R为离原子核的距离。简单的说就是呈6次方衰减。
结构优化使用的是软件默认的解析梯度方法。
超分子的结构优化是在气相中完成(在ADF软件中,创建超分子,并使用ADF模块进行计算,默认情况即为气相,也就是说超分子周围没有其他分子,边界在无穷远处)。使用Cs对称,即超分子为平面结构。结构优化完成之后,进行频率计算,没有发现虚频,从而确认是能量局域最小点(注:几何结构优化算法无法得到全局最小点,用一个比喻来说明:一个凹凸不平的地面,一个小珠子被放在某一个位置,那么它将自动滚入所在的“坑”里面,而无法滚入到海拔最低的坑里面。几何结构优化算法,类似于在重力作用下,珠子滚到低处的过程)。计算中并没有考虑BSSE(关于BSSE的原理和计算方法,可以具体参考费米维基:bsse),因为BSSE已经部分地被色散修正涵盖。
ADF软件操作
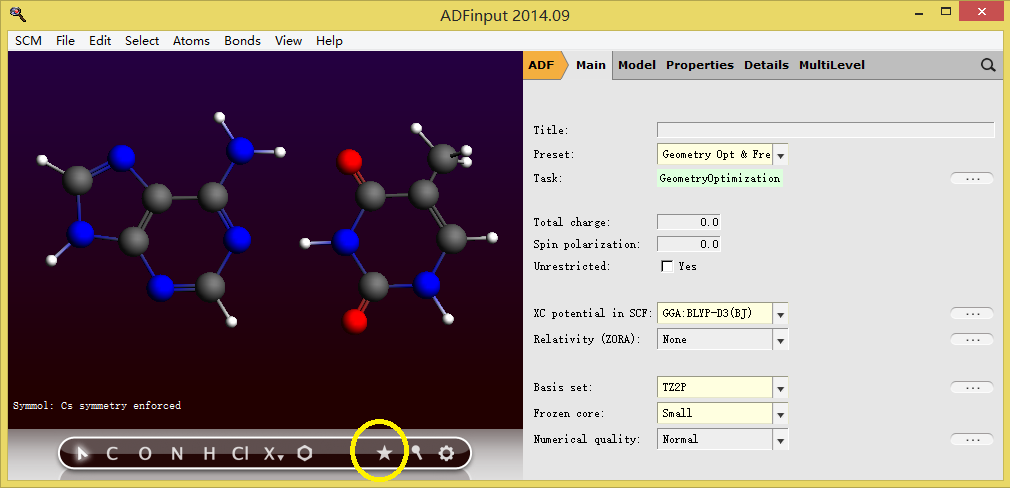
在ADF软件中,我们进行结构优化和频率计算的参数设置如下所示(注意运用了下图中黄色圆圈中所示的五角星工具,这个工具是自动对称化,也就是让分整个窗口内的分子体系,自动靠近最可能的对称性,本例中,手动将分子大致调整为平面结构,点击五角星工具之后,自动对称化为Cs点群):
ADF操作图示 1
键能分解(EDA)
其物理意义的详细讲解,参考能量分解分析(EDA)
电荷分布分析
结果与讨论
AT与类似模仿物的结构和稳定性
我们通过计算A和T及其类似模仿结构的所有可能组合,研究DNA碱基对的氢键中,π电子和芳香性的重要程度。氢键键长和能量使用BLYP-D3(BJ)/TZ2P计算。二聚体对称性设定为Cs(镜面对称),单体对称性设定为C1(没有对称性)。
ADF计算操作流程
建模的操作,参考:AMS软件建模教程
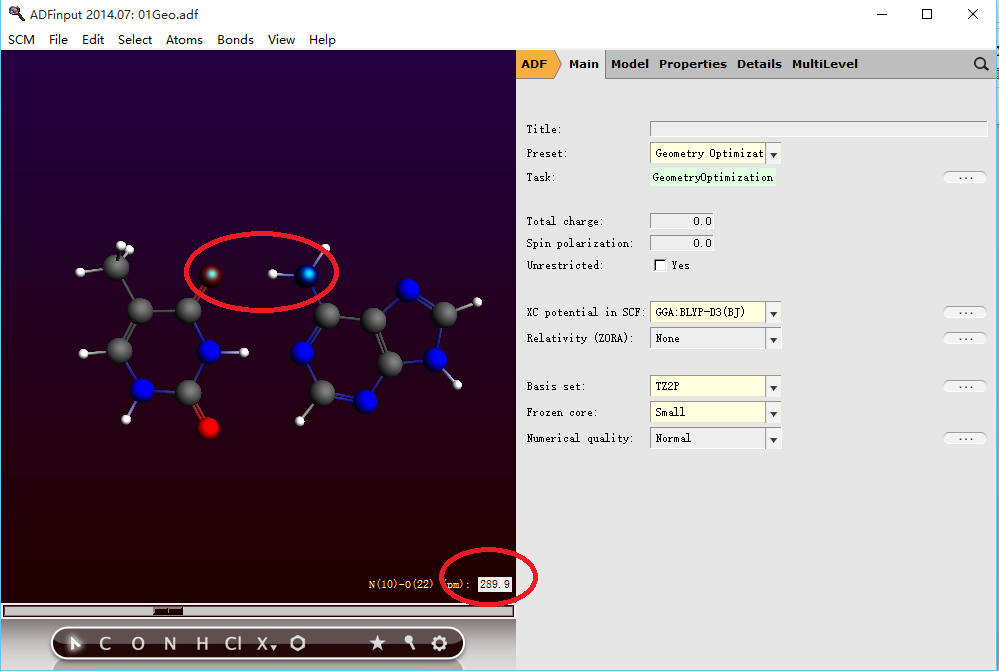
参数设置参考2.1节ADF操作图示1所示,如果能量震荡较为明显可以参考“费米维基:优化分子的几何结构”。注意如ADF操作图示1所示,将结构对称化为Cs群。结构优化任务文件名,此处命名为01Geo。产生的各种文件都自动以01Geo添加对应的后缀,例如日志文件名为01Geo.logfile。详见计算文件包下载链接。
优化完成后(在*.adf文件同级目录内,有了*.logfile和*.t21文件),在该任务的ADFinput窗口(或者ADFjobs窗口将结构优化任务这一栏目选中,变成灰色),SCM LOGO > Movie,则打开该任务结构优化动画。动画自动播放到最后一帧,然后点击File-update Geometry in ADFinput。如此最后一帧优化成功得到的分子结构将更新到ADFinput窗口。
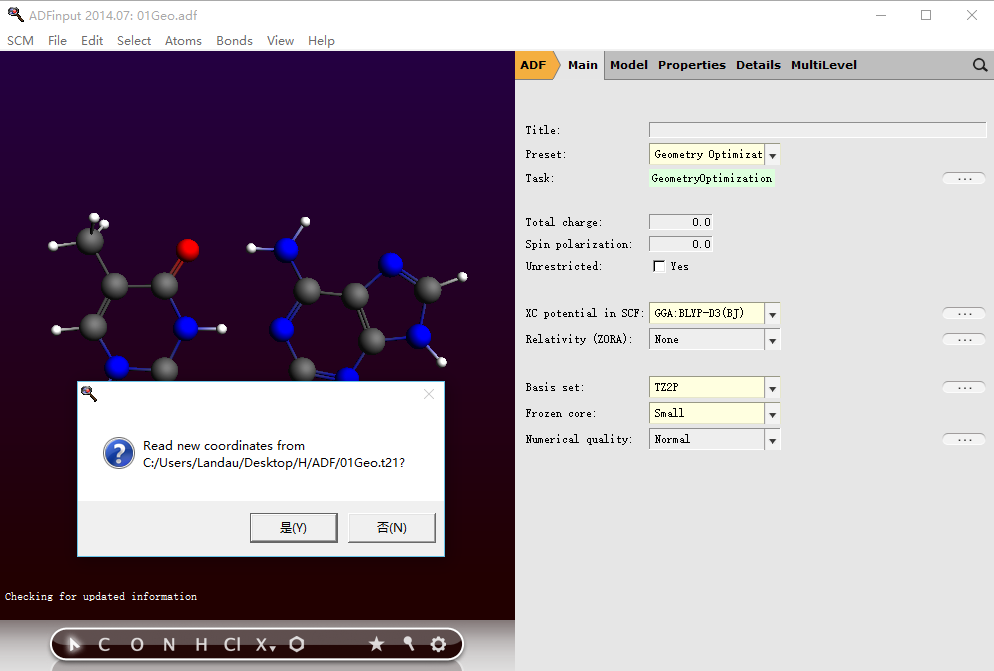
如果是纯粹的图形界面操作,也可以在优化完成后,ADFinput窗口自动弹出询问“是否将该窗口中分子结构更新为优化完成之后的结构”,选择“是”:
ADF操作图示 2
按住Shift键,选中要量取的两个原子:
ADF操作图示 3
显示两个原子距离为289.9pm也就是2.899埃;同样的操作,得到N-N距离为2.82埃。
文献中得到的结构,如下图所示(键能需要基于这个优化完成的结构另外计算单独计算,见接下来的内容):
图1 氢键键长(单位:埃)与能量(单位kcal/mol)
其中键长单位为埃,能量单位为kcal/mol。
接下来计算键能:
在第一步的ADFinput窗口中,修改任务参数如下:
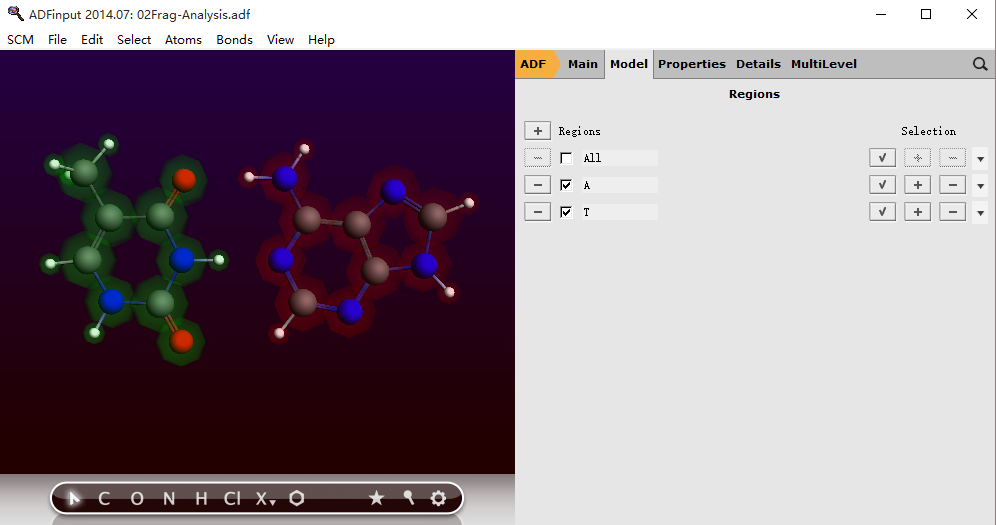
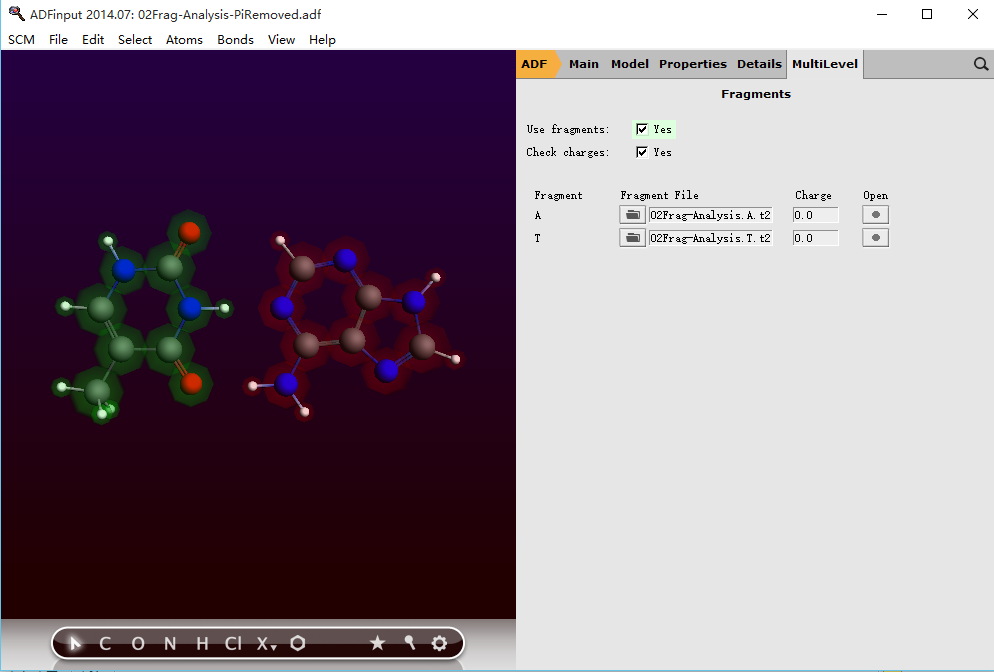
1)分子结构为前一步二聚体优化结束后得到的结构。此时不需要再对称化,软件将自动检测到对称性为Cs群。将该二聚体分为两个区,分区操作参考如何创建分区。
ADF操作图示 4
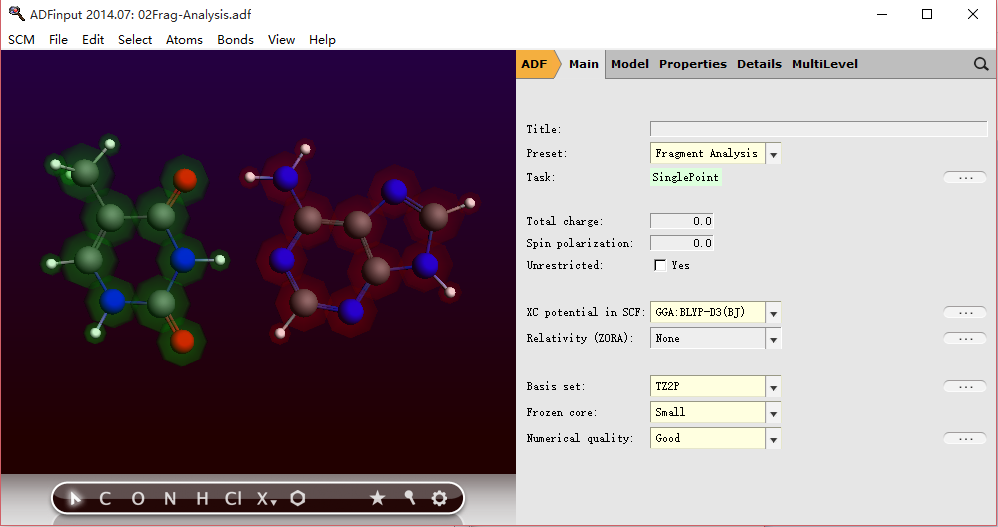
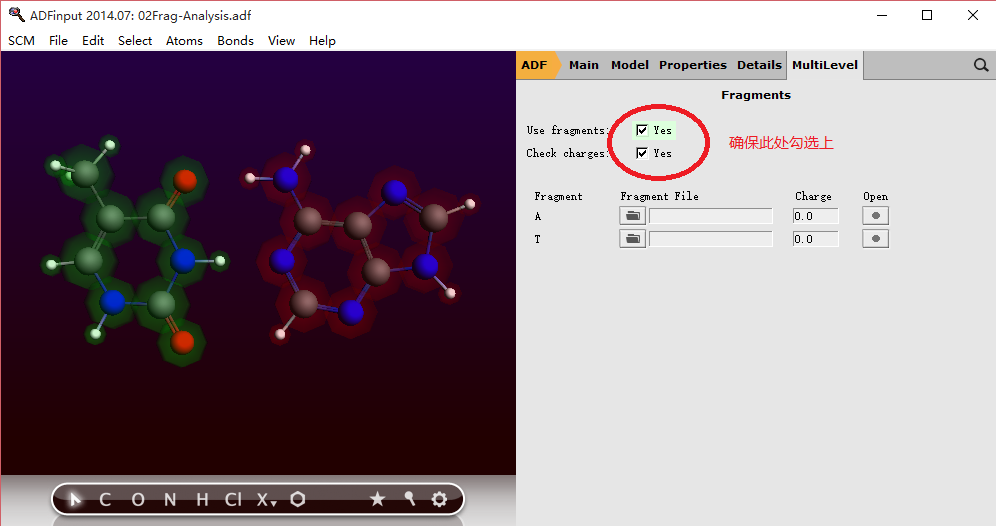
2)修改主面板参数:
ADF操作图示 5
ADF操作图示 6
3)File-save as保存为另外一个任务名,例如此处命名为02Frag-Analysis(注意见计算文件包下载链接)。如此将自动产生三个任务,即计算片段A的任务、计算片段T的任务、整个分子片段分析的任务。如果是在图形界面提交,则只需要File-run就可以了。如果是要提交到Linux服务器,提交方式参见:“正式版的安装、维护与升级”
在ADFinput窗口点击SCM LOGO > logfile查看结果单体之间的结合能:
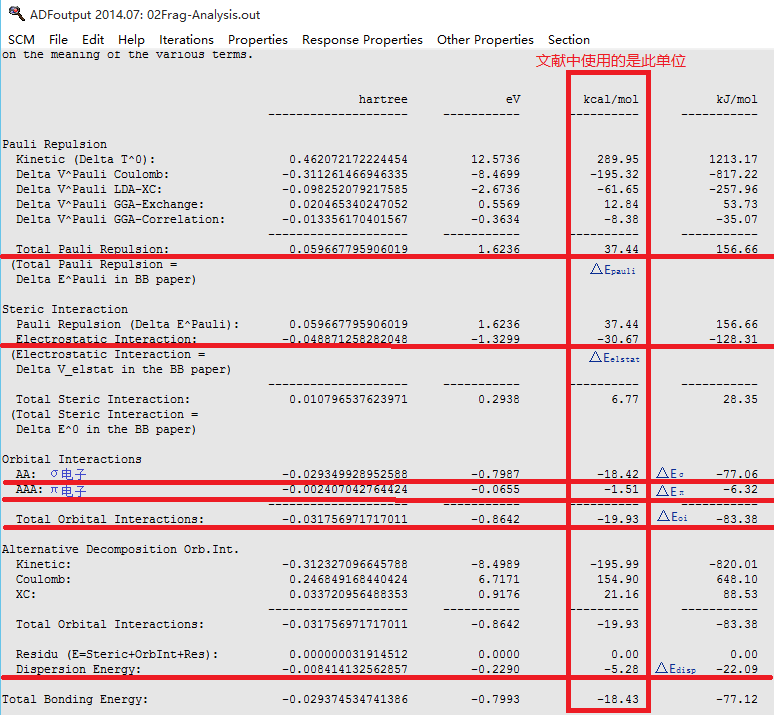
<Aug26-2015> <18:07:37> Bond Energy -18.43 kcal/mol
这是完成形变准备的单体结合在一起形成二聚体的键能,或者说释放的能量。即△E=△Eprep+△Eint中的△Eint。图1中的能量还包括形变准备能△Eprep。也就是自由状态下的两个单体和完成形变准备之后的单体之间的能量差(后者大于前者,因为前者是单体的能量最低点,而后者不是最低点),因此与-18.44kcal/mol符号相反,应为正值。
计算形变准备能
采用ADF操作图示1中完全相同的参数(另外按照文献的要求,将对称性取消,操作方式为:details-symmetry,将Auto改为Nonsym,另外如果前面设定了优化步长,这个时候也可以取消,details-Geometry convergence恢复默认设置就可以了——对于小分子,默认的优化算法效率高的多。两个计算任务均如此设置),但窗口左边的分子是A或者T单体——二者都需要计算一遍。任务保存为03A-Free、03T-Free。同样地查看其Bonding Energy(实际上就是总能量),分别为:-2308.22kcal/mol、-2168.75kcal/mol;
而上一步中,进行Fragment Analysis的时候,自动计算了已经完成形变准备的A和T的总能量,打开02Frag-Analysis.A.logfile和02Frag-Analysis.T.logfile可以分别看到二者的能量:-2307.65kcal/mol、-2167.50kcal/mol。
因此形A和T的变准备能分别为-2307.65-(-2308.22)=0.57kcal/mol,以及-2167.50-(-2168.75)=1.25kcal/mol,因此△Eprep=0.57+1.25=1.82kcal/mol。
因此,△E=△Eprep+△Eint=-18.43+1.82=16.61kcal/mol。对应的是图1中AT第三个的数据(来自文献)为-16.7kcal/mol。
从计算结果来看,不同大小的芳香系统或者不同数目的π电子,氢键的键能在-16.8kcal/mol到-15.2kcal/mol之间,变化不大。最大的AT与最小的A’’T’’,氢键的键能只相差0.6kcal/mol。随着π电子的改变,氢键键长的变化似乎显著一些(大于0.01埃。注:原文为0.1埃,不清楚为何)。对某个相同的配体,A、A’、A’’三者氢键键长相似;对相同的配体,T、T’、T’’三者变化规律不同:对于T和T’,N6(H)-O4的距离比N1-(H)N3的距离大0.05-0.08埃;但对于T’’,变成前者比后者小0.02-0.06埃(注:原文此处误作“0.2-0.6埃”)。
氢键相互作用的本质
下一部分,我们将讨论AT及其模仿类似结构氢键的本质。之前的工作[5a],阐明了Watson-Crick碱基对AT和GC中,静电与共价相互作用对成键机制的影响。
A、A’、A’’与T、T’、T’’对电子结构的影响
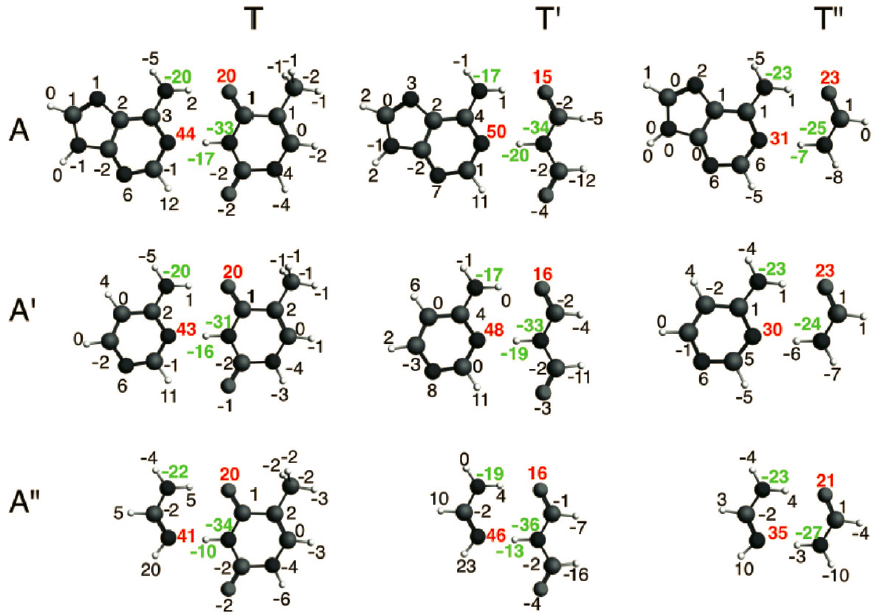
之前,我们已经展示了A和T电子结构方面有互补的特性,也就是说,接收质子的原子是负电性的,而质子本身带上了正电。A和T的小类似模仿结构也是如此(从下图可以看出)。对于A、A’、A’’,N1和H6的带电量差别不大,对于T、T’、T’’,H3和O4带电量的变化也不大(前面已经提到,氢键键能、键长的差别也不大)。
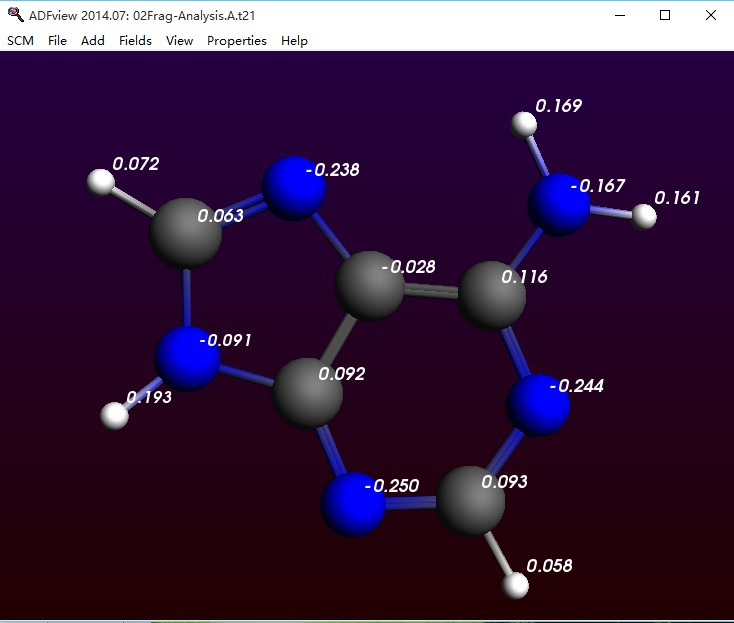
图2 完成“形变准备”单体的VDD电荷(单位me-)
VDD电荷计算的ADF软件操作:
上图所示的VDD电荷是指完成形变准备的单体相对于孤立原子的电荷变化。因此查看完成形变准备的单体的VDD就可以得到上面的信息。而02Frag-Analysis这个任务自动包含三个任务:02Frag-Analysis,以及02Frag-Analysis.A、02Frag-Analysis.T。后面这两个任务实际上就是完成形变准备的A、T单体的计算。因此我们查看该任务的结果就可以了:
SCM LOGO > jobs,弹出任务列表菜单,选中02Frag-Analysis.A这一行(点击该行的任意位置均可选中该行),选中后变为灰色,然后SCM LOGO > View弹出该任务的图形化结果显示窗口,然后在View窗口点击Properties-Atom Info-VDD charge-show(对于片段而言,因为参考点是中性原子,因此这个数字相当于片段中每个原子此刻的净电量),即显示A单体的VDD电荷单位为e-(是文献图2中单位的1000倍):
ADF操作图示 7
其他单体情况可以类似的方式得到,在此不一一重复。
接下来我们考虑σ体系电荷转移相互作用的可能性。下图展示了DNA碱基A和T的施主-受主轨道相互作用:接收质子的N或者O的孤对电子,指向,并贡献电荷到另一个碱基的N-H基团的σ*空轨道上,这导致了非常弱的共价键(σLP+σ*N-H)的形成。对于氢键的共价成分的完整描述参考文献[5a]。
图3 A和T之间,N6(H)…O4和N1…(H)N3中最显著的(σ电子)施主-受主相互作用分子轨道图
对于不同的二聚体的同一种的施主-受主相互作用,A和T的较小类似模仿结构在σ电子体系中,需要具有相同的前线分子轨道。图4所示为A、A’、A’’以及T、T’、T’’的前线轨道图。这些轨道,除A’’和T’’的之σHOMO-1外(各自都只有一个接受质子的N 或O,因此只有一个孤对特征轨道),都非常相似。施主-受主相互作用发生于A(A’、A’’)的σHOMO-1和σHOMO,以及T(T’、T’’)的σLUMO+1或σLUMO,以及A(A’、A’’)的σLUMO和σLUMO+1。注意:对于N …(H)N氢键,A’’施主-受主相互作用只发生于A’’的σHOMO和T、T’、T’’的σLUMO+1或σLUMO之间;对于N(H)…O氢键,T’’施主-受主相互作用只发生于T’’的σHOMO和A、A’、A’’的σLUMO+1或σLUMO之间。
图3所示信息对应的ADF软件操作(使用ETS-NOCV功能,通过分析NOCV的电子转移情况,可以更容易得到图3信息,具体可以参考ETS-NOCV理论、EDA-NOCV化学键分析(分子间作用):二聚体能量分解(EDA)中轨道作用、电子在片段轨道间转移的分解):
注意:这个图与ADF操作图示8虽然恨接近,事实上,也是经由该图衍生的到的信息。不过并不是直接就是该图。事实是,图3所示的,是轨道相互作用部分,而轨道相互作用是对应着电子的转移(从一个片段的占据轨道转移到另一个空轨道),因此,实际上这个图对应的是表1的内容。也就是:
- A的HOMO失去电子,主要跑到T的LUMO+1,换成轨道相互作用的语言来描述,就是:A的HOMO和T的LUMO+1相互作用,A的HOMO实际上对应的就是AT的HOMO,T的LUMO+1对应AT的LUMO+2;也就得到了上面的图3;
- T的HOMO失去电子,主要跑到A的LUMO(以及LUMO+1),就是:A的LUMO(以及LUMO+1)与T的HOMO相互作用,T的HOMO实际上对应的是AT的HOMO-1,A的LUMO对应AT的LUMO+1
这些信息,详细地可以从下面的过程中得到:
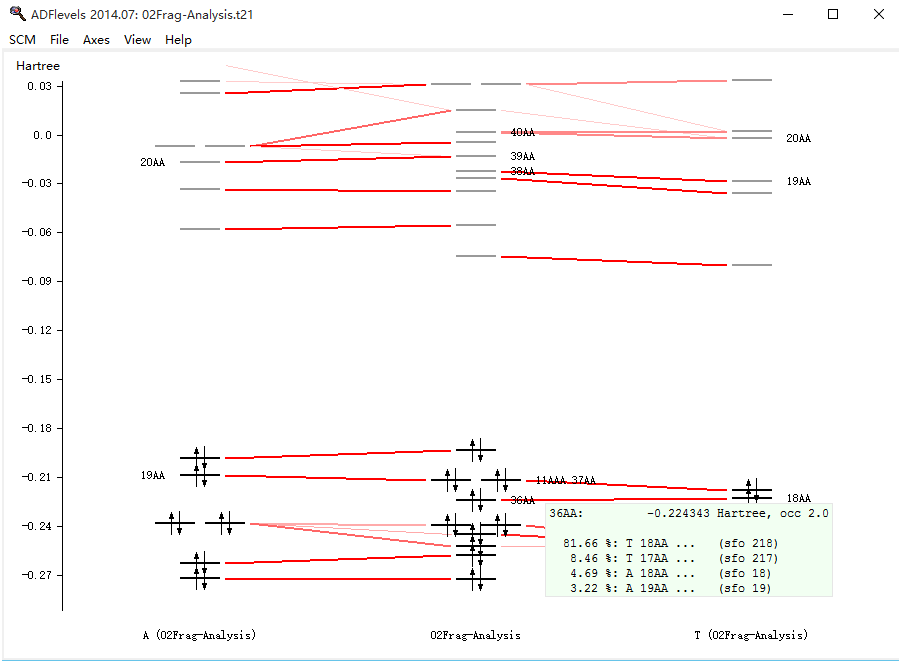
在adfjobs窗口,选中02Frag-Analysis这一行,点击SCM LOGO > level,则显示与图3类似的二聚体与单体之间的轨道关系图。中间为二聚体轨道、两边分别为单体A、T的轨道。鼠标放置在对应横线上则显示该轨道的不可约表示(对于Cs群,只有两种不可约表示AA和AAA,前者即σ,后者即π)、序号(按能量大小顺序排序,序号越大,能量越高)。但与图3不同的是,该图(ADF操作图示 8)显示了完整的单体与二聚体轨道关系,而图3只列举了σ电子中少数几个轨道的关系,并且只显示了其中施主、受主效应相关的轨道关系。
ADF操作图示 8
与图3对应的,左边A(02Fragment-Analysis)上方的20AA就是图3中Adenine的σ的LUMO,19AA即Adenine的σ的HOMO;02Fragment-Analysis上方的36AA、37AA即图3中AT的σ的HOMO-1和HOMO,更上方的三条38AA、39AA、40AA以此类推对应图3中AT上方的三个空轨道;T(02Fragment-Analysis)与此类似。
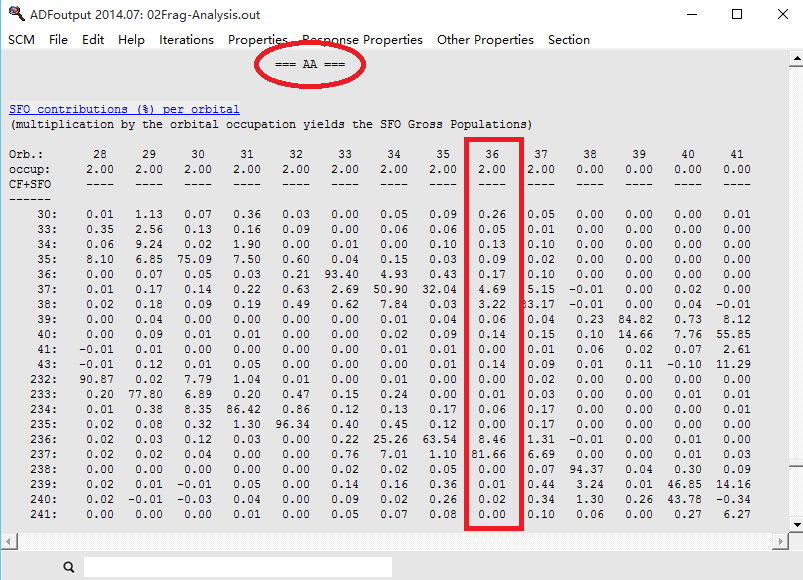
当鼠标放置在ADF操作图示 8中02Fragment-Analysis上方某条轨道上,即会如图中所示,显示该二聚体轨道的组分的主要来源。例如图中所示为36AA的组分来源于T的18AA,这与图3是一致的。另外有一些其他小组分的A和T的占据轨道,这些小组分和电子施主-受主的效应没有关系,那么要查看该轨道的组分的详细来源,就需要到out文件里面去核对,out文件里面的数据详细的多。在level窗口,点击SCM LOGO > output,我们在新的out窗口,点击Properties-%SFO per orbital,看到如下内容:
ADF操作图示 9
“ADF操作图示 8”与“ADF操作图示 9”可以对照起来看。后者红色圆圈表示下面的内容是AA不可约表示,也就是σ电子轨道的信息。方框里面36表示36AA,下面的2表示该轨道占据了2个电子。更下方的数据,则是该轨道的详细的组分来源(百分比)。例如81.66也就是“ADF操作图示 8”中T单体的18AA轨道(HOMO),而该单体轨道(最右边的一列数字)在此处的编号为237(同一个单体的轨道的编号是连续的),因此“ADF操作图示 8”中的T单体的19AA、20AA分别对应此处编号,分别即为238(LUMO)、239(LUMO+1)。类似的,“ADF操作图示 8”中占3.22%的是A单体的19AA,在此处的编号为38(HOMO),那么对应的A单体20AA(LUMO)、18AA(HOMO-1)在此处的编号应为39(LUMO)、37(HOMO-1)。
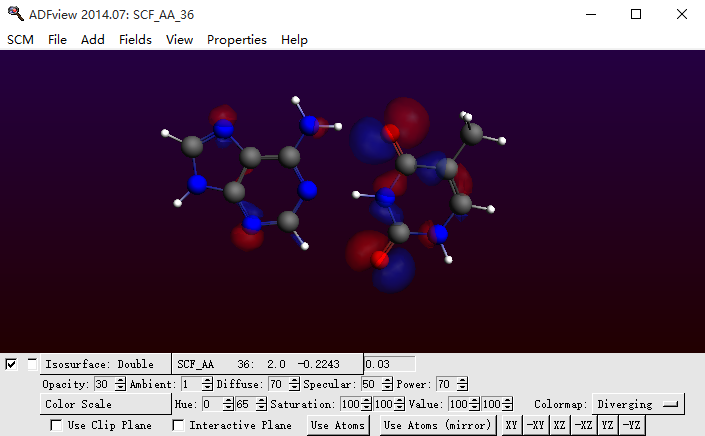
那么跟电子施主-受主效应相关的是空轨道的组分(即序号大于38,以及大于237的单体轨道),因此我们查看一下“ADF操作图示 9”中,AT的σ轨道HOMO-1贡献较大的空轨道:39、40、43——也就是A单体的20AA、21AA、24AA(即LUMO、LUMO+1、LUMO+4)。与图3略有差别:图3中主要来自A单体的LUMO。在level窗口查看AT的σ轨道HOMO-1该轨道的空间分布(右键点击该轨道的短横线,选择36AA,则弹出36AA的轨道空间分布):
ADF操作图示 10
该轨道实际上就是对应图3中的N6(H)…O4氢键,从上面的分析可以得出电子从T转移到A的空轨道上面去了,转移量很小(本节下文中的表1分析了AT的电子转移,大约0.01电子的数量级,从T转移到A)。这与图3的结果是一致的。
类似地,AT的σ轨道HOMO贡献较大的空轨道:239、240——也就是T单体的LUMO+1和LUMO+2。与图3一致:来自LUMO+1。对应N1…(H)N3氢键。
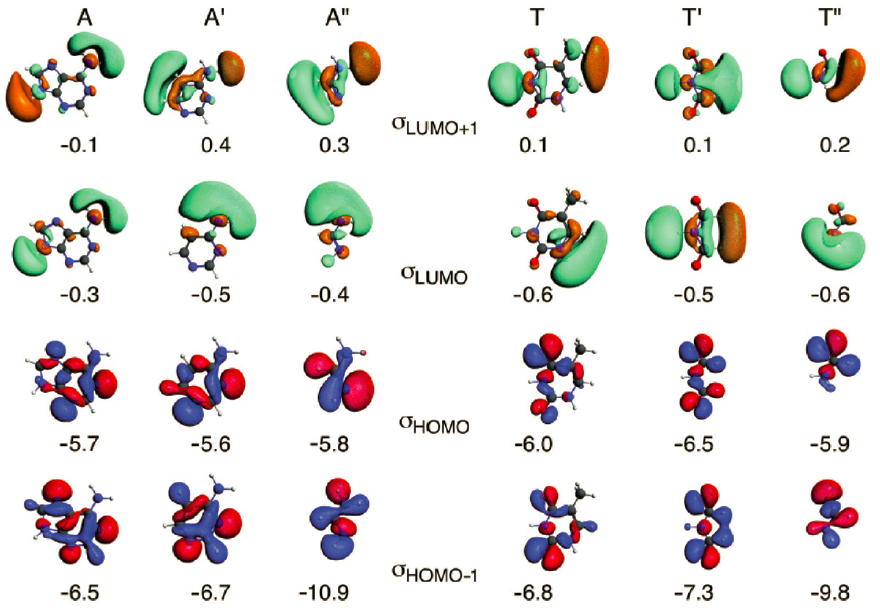
在ADFjobs中选中02Fragment-Analysis.A,之后点击SCM LOGO > level,然后用上面的方法,可以分别得到完成形变准备的A的HOMO-1、HOMO、LUMO、LUMO+1轨道的空间分布,与文献中图4所示是一致的。不同的颜色表示相位相反。
图4 单体的σ电子前线轨道(HOMO-1,HOMO,LUMO,LUMO+1)及其对于能量(eV)
单体形成二聚体时,σ电子的施主-受主相互作用,带来σ占据轨道的占据数微微的减小,这些电子跑到σ电子的空轨道上面。在表1中,列出了不同单体σHOMO-1,σHOMO,σLUMO和σLUMO+1的总占据数。总占据数是从完成“形变准备”的单体结合形成二聚体的计算中得到的。即:按二聚体分子轨道中单体轨道σHOMO-1,σHOMO,σLUMO和σLUMO+1的权重得到,这是因为二聚体分子轨道由单体轨道做基函数展开得到。总占据数很小(小于0.10个电子),这与之前的结果[5a]是一致的。其他轨道例如σHOMO-2或σHOMO-3也发生占据数少量减小,或σLUMO+2和σLUMO+3的占据数发生少量增加。这些不是主要的相互作用,因此表中未列出。
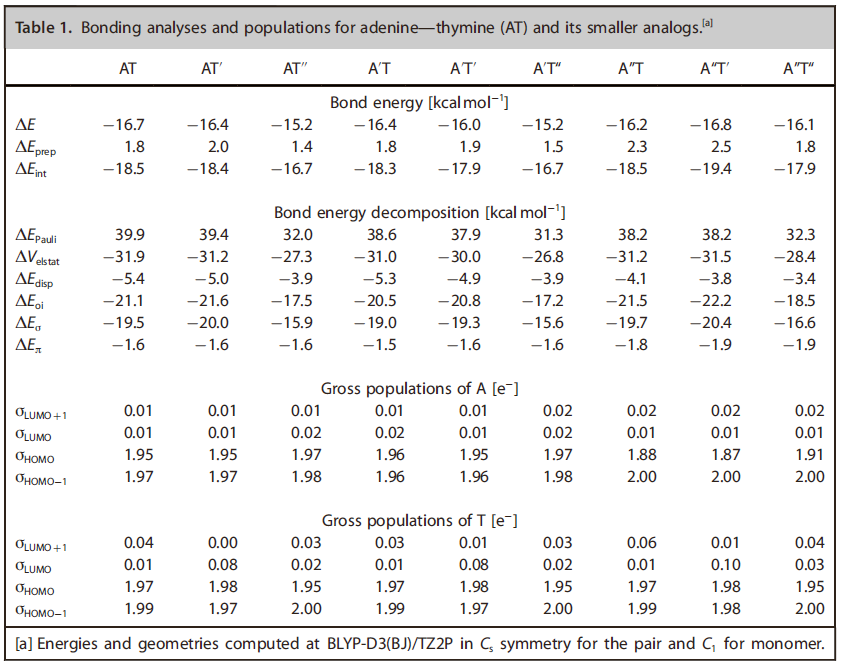
表1 键能分析、AT及其类似物布居分析
以AT为例,表1的数据从ADF的计算结果中找到:
△E、△Eprep、△Eint在3.1节中已经讨论过了;下面的6个能量如下图(在ADFjobs窗口选中02 Fragment-Analysis ,点击SCM LOGO > Output打开out文件窗口,之后点击Properties-Bonding Energy Decomposition)所示:
ADF操作图示 11
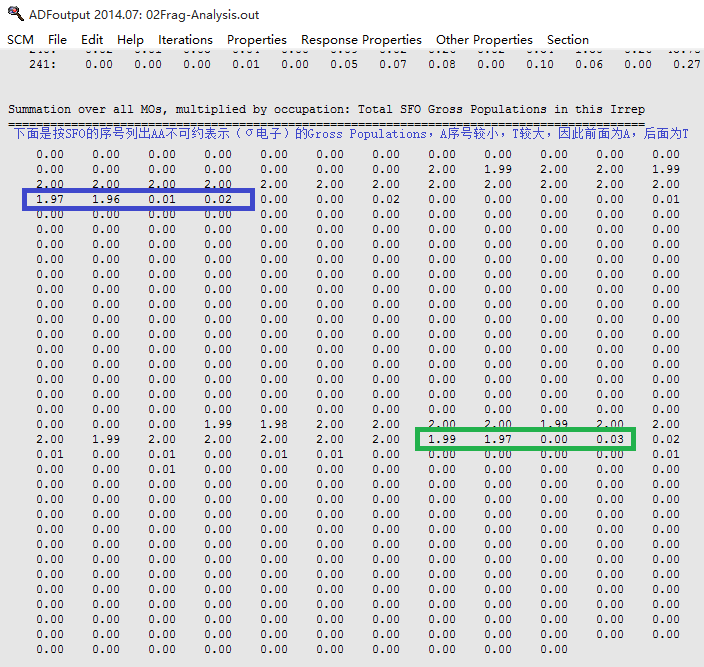
点击Properties-%SFO per orbital,往下翻半页即见到如下图所示的内容(AA不可约表示,也就是σ电子的Gross Populations;再往下翻即为AAA不可约表示,也就是π电子的SFO Contribution (%) per orbital,及其Gross Populations):
ADF操作图示 12
蓝色框的四个数字分别为A的HOMO-1、HOMO、LUMO、LUMO+1的Gross Populations;绿色框的是T的HOMO-1、HOMO、LUMO、LUMO+1的Gross Populations。与表1略有差别,但定性结论不变。
氢键能的定量分解
前一部分我们确定了A和T的类似模仿结构的电荷分布正好适合互相吸引。在之前的工作[5a]中,已经阐明了σ电荷转移以及π电子的极化。我们希望和Watson–Crick碱基对AT一样定量地估算二聚能量的各个组分大小。因此我们对不同的二聚体进行了键能分解(表1)。
键能首先分解为单体的“形变准备”能△Eprep以及单体之间的相互作用能△Eint,前者非常小(1.4到2.5kcal/mol),这是由于氢键导致的形变量很小所致。△Eint的变化趋势与键能相同。△Eint的变化量大的多,介于-16.7kcal/mol到-19.4kcal/mol之间。
进一步分解△Eint发现,对于所有的情况, 静电作用△Velstat都不提供净的键能,它只能部分补偿Pauli排斥轨道相互作用△EPauli。(在附录的AT的计算文件中,我们看到△EPauli和△Eelstat分别为37.44kcal/mol和-30.67kcal/mol,见ADF操作图示 13)因此,如果没有键轨道之间的相互作用能△Eoi,单体将会互相排斥。而轨道相互作用又分为σ组分和π组分。△Eσ是上面提到的电子施主-受主相互作用的主体。△Eπ是导致极化的主因,这又部分地消减了由σ电子的电荷转移导致的局域的电荷堆积(见3.3 电荷重新分布)。色散项描述色散相互作用修正,在-3.8kcal/mol到-5.4kcal/mol之间。
ADF操作图示 13
σ轨道相互作用相△Eσ是两个氢键的施主-受主相互作用的总和。为了得到N(H)…O和N…(H)N的施主-受主相互作用各自大小的定量估计,我们去掉π空轨道,如此就“人为地”关闭了π电子引起的极化效应。然后去掉其中一个碱基的σ空轨道来人为地关闭对应的氢键的施主-受主相互作用。对A、T及其类似模仿物进行了相同的处理过程。这样氢键的σ相互作用△Eσ(σ,-; σ,-)就是在没有π极化的干扰下进行分析。对比表1的△Eσ和图5中的△Eσ(σ,-; σ,- ),结果表明,在π极化参与的情况下,施主-受主相互作用只低0.3kcal/mol。因此我们能够得出结论(对于A和T的类似模仿结构也成立):σ和π协同作用很小。图5也列出了每个氢键的施主-受主相互作用:N(H)…O的△Eσ(σ,-;-,-)去掉了T、T’、T’’的σ空轨道;N…(H)N的△Eσ(-,-;σ,-)去掉了A、A’、A’’的σ空轨道。
通过对比△Eσ(σ,-; σ,- )与△Eσ(σ,-;-,-)+△Eσ(-,-;σ,-),可以弄清σ电子体系中,电荷从一个碱基通过氢键转移到另一个碱基与另一个碱基通过另外那个氢键转移回来,二者之间的协同作用。这些值表明,两个氢键各自独立地反方向转移电荷,这与与我们之前发表的工作是一致的。另外,对于T、T’相关的二聚体,N…(H)N氢键的强度是N(H)…O的二倍;但对于T’’相关的二聚体,两个氢键强度相当。
图5 总的σ相互作用△Eσ(σ,-; σ,- )与N(H)•••O的σ相互作用△Eσ(σ,-;-,-)和N•••(H)N的σ相互作用△Eσ(-,-;σ,-)
对应的ADF计算操作:
ADF可以通过关键字设置删除掉某些片段轨道,以此删除分子的空轨道:
REMOVEFRAGORBITALS Fragtype(片段的名字) subspecies nremove(不可约表示符号,以及该不可约表示需要删除的轨道数目) subspecies nremove ... subend fragtype subspecies nremove ... subend .... end
其中fragtype是片段名称(即Model-Regeon里面为片段取的名字),subspecies 是该片段的轨道不可约表示的标记,nremove是该不可约表示下需要删除的能量最高的轨道的个数。
以下分别演示:
- 去掉两个碱基的π空轨道,没有π极化参与的情况下的计算设置与结果;
- 去掉A的σ空轨道以及两个碱基的π空轨道,得到N…(H)N的键能计算设置与结果。
如下第一个范例:
去掉两个单体的π空轨道,计算σ和π的协同效应有多大
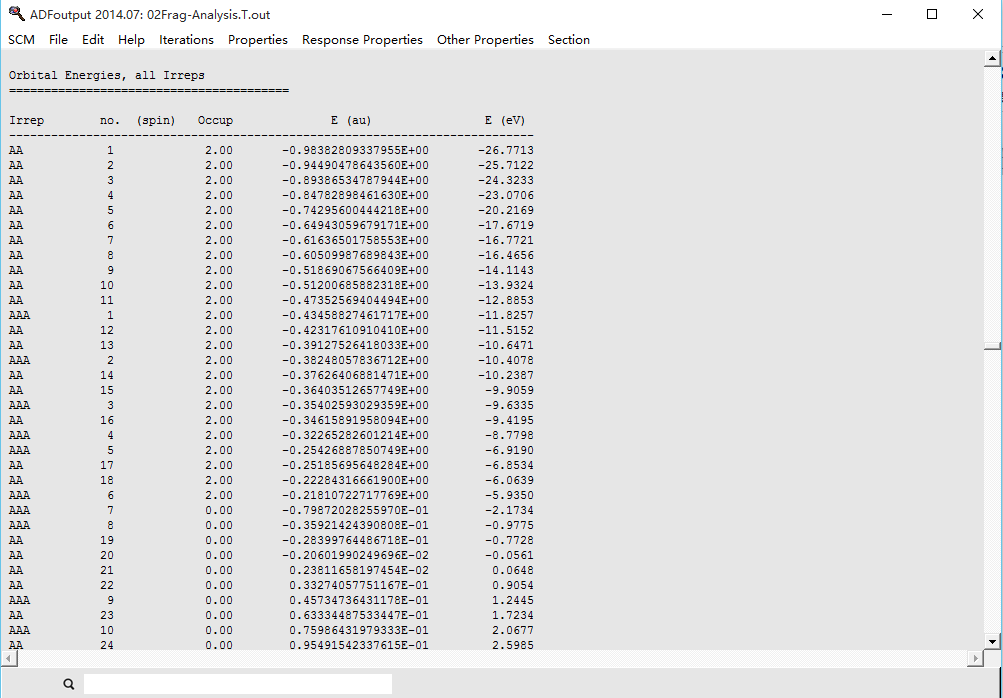
我们检查没有原始的计算结果中π空轨道的个数——在adfjobs中打开02Frag-Analysis.T和02Frag-Analysis.A的out(在ADFjobs窗口,选中02Frag-Analysis.T或02Frag-Analysis.A这一栏,然后点击SCM LOGO > Output),查看对应的单体的所有不可约表示的所有轨道的列表:
ADF操作图示 14
其中第一列是不可约表示符号,AA表示σ,AAA表示π;第二列是在该不可约表示内,轨道能量从低到高的序号;第三列是该轨道的电子占据数;第四列是该轨道的能量,单位为Hartree;第五列是该轨道能量,单位为eV。因此我们找到AAA不可约表示里面,占据数为2的序号最大的是6,也就是AAA的前6个轨道是占据的,后面的都是空的,再看AAA的最大序号是95,因此T的π空轨道有95-6=89个。顺便我们得到σ空轨道为187-18=169,用于第二个范例。
类似地我们可以知道A的π空轨道也有89个,σ空轨道为181个。
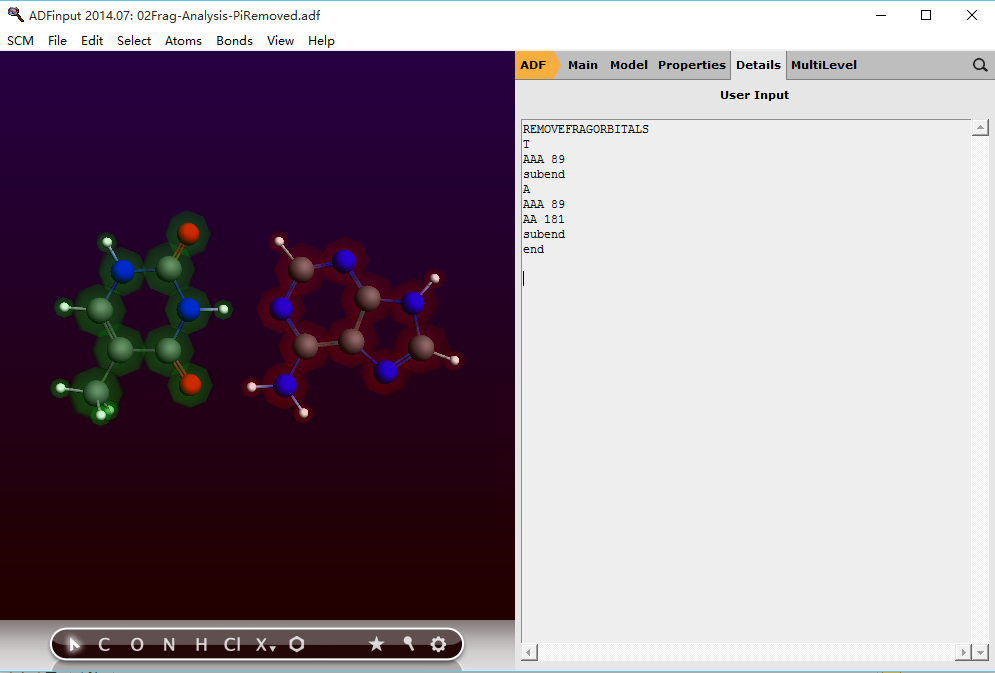
在02Frag-Analysis的基础上,增加如下设置,以计算去掉两个碱基的π空轨道的结果。下图中Details-Userinput中插入的内容为:
REMOVEFRAGORBITALS T AAA 89 subend A AAA 89 subend end
ADF操作图示 15
注意:片段名字只取后面一段就可以了,以Model-Region里面的名字为准。
片段A、T因为前面已经计算过了,此处就不需要再重新计算了,因此直接读入前面计算生成的*.t21文件即可:
ADF操作图示 16
之后,文件另存为02Frag-Analysis-PiRemoved.adf,然后运行此任务。运行完毕之后,查看out中Properties-Bonding Energy Decomposition:
Orbital Interactions AA: -0.029056837590716 -0.7907 -18.23 -76.29 AAA: 0.000000000000002 0.0000 0.00 0.00
可以看到Eoi中AAA(π)的贡献已经被清理出去了;σ部分为-18.23kcal/mol(对应图5中AT中-19.3),原始的没有清理π空轨道的结果为-18.42kcal/mol。σ部分的Orbital Interactions主要是施主-受主作用,π对它的影响仅为0.2kcal/mol(文献中给出的数值为0.3kcal/mol),因此得道二者的协同效应非常微弱。
下面进行第二个范例:
去掉A的σ空轨道以及两个碱基的π空轨道,得到N…(H)N氢键能
同样地,在02Frag-Analysis-PiRemoved的基础上,增加如ADF操作图示 17所示设置。图中Details-Userinput中插入的内容为:
REMOVEFRAGORBITALS T AAA 89 subend A AAA 89 AA 181 subend end
ADF操作图示 17
另存任务名为02N-HN.adf,并运行之。查看out中Properties-Bonding Energy Decomposition:
AA(σ)的Orbital Interactions为-11.94kcal/mol(文献中如图5中给出的数值是-12.6kcal/mol)。 类似地,可以计算得到N(H)…O氢键的AA(σ)的Orbital Interactions为-7.26kcal/mol(文献中如图5中给出的数值是-7.6kcal/mol),对应的输入文件为02NH-O.adf。
氢键引起电荷重新分布
到现在为止,我们的研究已经表明A和T的较小同类模仿结构与AT二聚体的成键特征相同。这表示,这些氢键中,参与成键的原子并不需要与芳香环连接来达到氢键目前的强度。在这一节中,我们研究氢键的形成,导致的σ和π电子的重新分布。因此我们使将VDD原子电荷划分成σ和π两个组分(见2.3节中的方程)。对于Pauli排斥引起的电荷重新分布,见本文的Supporting Information。
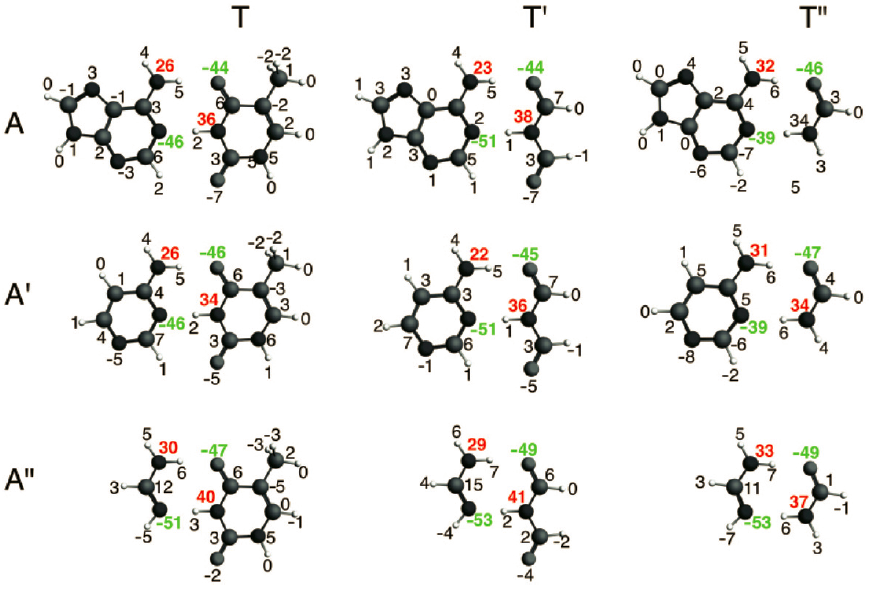
图6 各个结构的△QσA,oi的贡献(σ电子的贡献)
图7 各个结构的△QπA,oi的贡献(π电子的贡献)
图6、图7分别描述了九个体系的σ和π电荷重排。△QσA,oi显示出AT及其类似模仿结构的清晰的电荷转移:在电子施主原子上负电荷丢失,但在接收电子的N-H键的N原子上,有明显的负电荷堆积(见图6)。π电子密度极化方式为:σ电子的电荷转移导致的电荷堆积被抵消、补偿——电子施主原子得到π电子密度,N-H键中接收电子的N原子失去π电子密度(对比图6、7中的△QσA,oi和△QπA,oi)。π电子密度的这种重排方式与Gilli等[3]建议的Lewis结构是一致的。T’’的电荷重排要比T和T’小一些,T’’的轨道相互作用也要弱一些,这两点是一致的。后者应该是前者主要原因。更进一步,我们看到σ和π电荷重排不依赖于芳香环。
图6和图7,使用ADF软件计算如下:
△QσA,oi和△QπA,oi分别是σ(AA不可约表示)、π(AAA不可约表示)的贡献,按照2.3节中的定义,二聚体中每个原子的VDD电荷是以片段为参考的,因此在计算上,仍然使用与02Frag-Analysis完全一样的设置,但有2个区别:
1)因为单体已经计算过了,所以此时不用再计算了,在MultiLevel-Fragment中读取之前单体的*.t21文件。这样可以省掉计算单体的时间。
ADF操作图示 18
2)需要先计算去掉σ(AA不可约表示)、π(AAA不可约表示)所有不可约表示的空轨道,Details-Userinput输入如下:
REMOVEFRAGORBITALS T AAA 89 AA 169 subend A AAA 89 AA 181 subend end
之后在View里面查看properties-Atom info-Voronoi Charge (SCF),如此显示每个原子的VDD电荷(这个值,实际上就是纯粹Pauli作用引起的VDD电荷)。比较方便的做法,是先选中关心的原子,然后再按上述的方式显示VDD(SCF),如此就只显示预先选定的原子的VDD(SCF),其他原子的Voronoi Charge(SCF)就不显示了。原子的其他性质,也可以类似地只显示选定原子的。
3)分别计算只去掉σ(AA不可约表示)空轨道,或者π(AAA不可约表示)空轨道的Voronoi Charge(SCF),参数设置与2相同,唯一差别是Details-Userinput输入如下:
或者
Details-Userinput输入如下:
REMOVEFRAGORBITALS T AA 169 subend A AA 181 subend end
或者
REMOVEFRAGORBITALS T AAA 89 subend A AAA 89 subend end
如此就分别去掉了σ(AA不可约表示)空轨道,或者π(AAA不可约表示)空轨道,也就是,分别只保留了π对Voronoi Charge(SCF)的贡献或者σ对Voronoi Charge(SCF)的贡献。
4)用3)中得到的Voronoi Charge(SCF)减去2)中得到的纯粹的Pauli对Voronoi Charge(SCF)的贡献,差额就是纯的σ或π对Voronoi Charge(SCF)的贡献。也就对应了图6、7中的△QσA,oi和△QπA,oi。具体数字与文献有较大差异,但趋势不变、结论不变,并且△QσA,oi和△QπA,oi总和与文献一致。
sp2杂化还是sp3杂化?
在之前的章节,我们已经明确了π电子数目不会影响氢键能;也就是说,AT系列二聚体的氢键强度,彼此之间只相差1.6kcal/mol。这导致如下结论:虽然质子施主和受主原子并不需要是芳香环的一部分来加强氢键——但,sp2杂化是必须的吗?如果是,那么sp2杂化对氢键的强化能力有多大?或者说让氢键中质子施主和受主之间的距离减小了多少?[3]
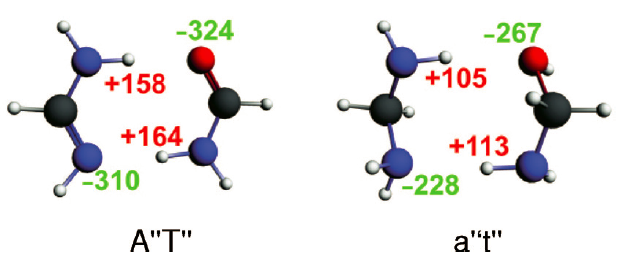
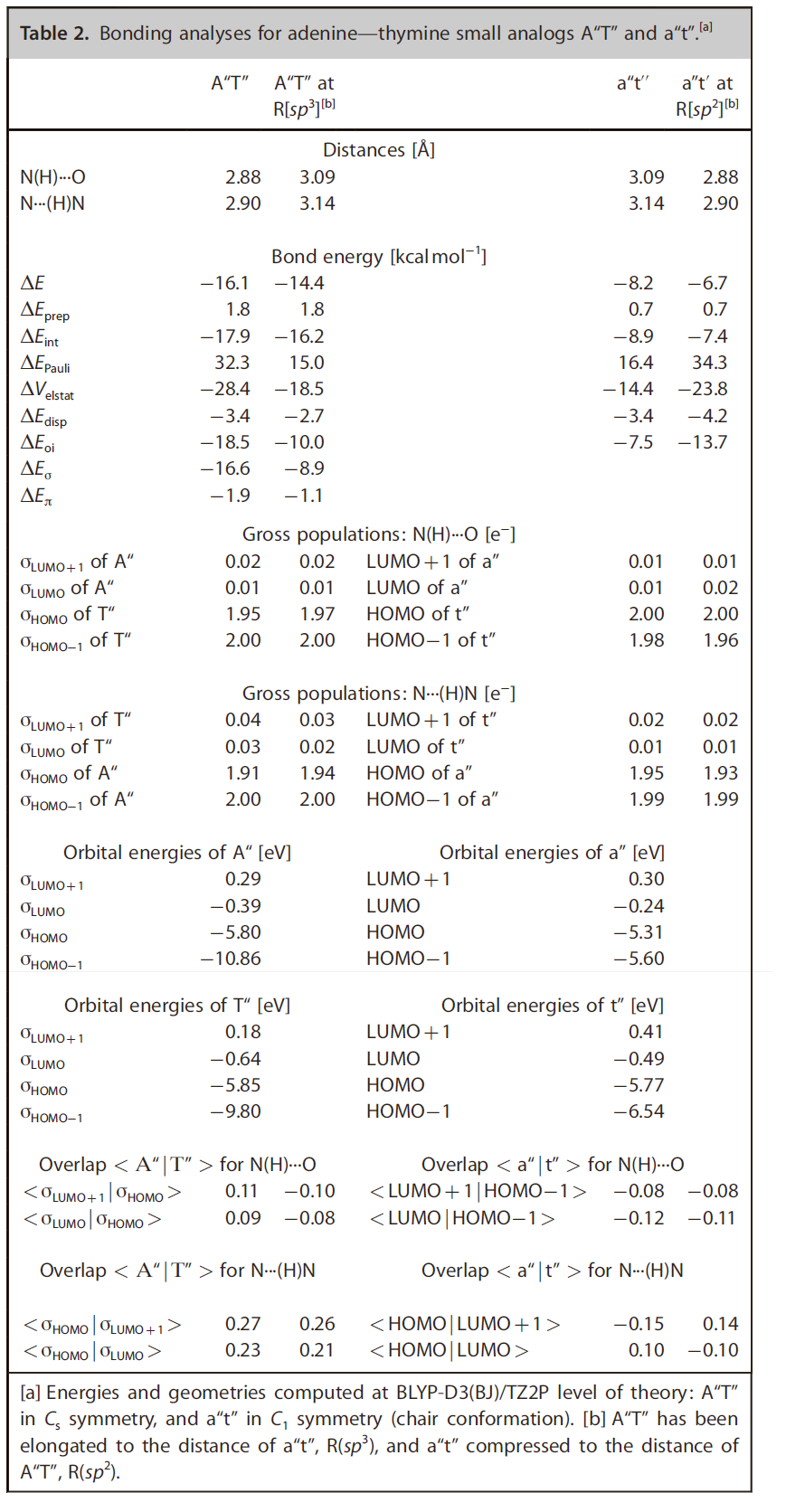
本节将通过对比A’’T’’和a’’t’’(图8)阐明sp2杂化是否是必要的。a’’t’’存在船式和椅式同分异构体,和环己烷相似,椅式构象能量低4.8kcal/mol(见该文Supporting Information)。sp2杂化的A’’T’’氢键能比a’’t’’强7.9kcal/mol(表2)。这跟π电子无关,因为sp2杂化的A’’T’’极化仅仅为-1.9kcal/mol(见表1)。在平衡结构时(即稳定结构,或者说能量最低的结构),a’’t’’的相互作用能的所有成键组分,△Eoi和△Eelstat’,都比A’’T’’小。但Pauli排斥二者都很小。
为了分析不同氢化情况带来的差别的根源,我们将a’’t’’的氢键距离调整到和A’’T’’氢键距离R(sp2)相同;另外也将A’’T’’的氢键距离调整到与a’’t’’氢键距离R(sp3)一致。在R(sp2)的情况下,A’’T’’和a’’t’’的相互作用能分别为-17.9kcal/mol和-7.4kal/mol。对比在R(sp2)的情况下两种二聚体相互作用能的组分,发现静电相互作用和轨道相互作用比较大;二者的Pauli排斥几乎一样大(A’’T’’为32.3,a’’t’’为34.3)。A’’T’’的静电相互作用以及轨道相互作用分别比a’’t’’强4.6、4.8kcal/mol。π共振仅为-1.9kcal/mol(表2)。a’’t’’静电相互作用比A’’T’’小,本文认为是由于图8所示的Voronoi形变密度电荷的补偿所致。a’’t’’的VDD电荷比A’’T’’小。
图8 A’’T’’和a’’t’’前线原子的Voronoi形变密度VDD电荷
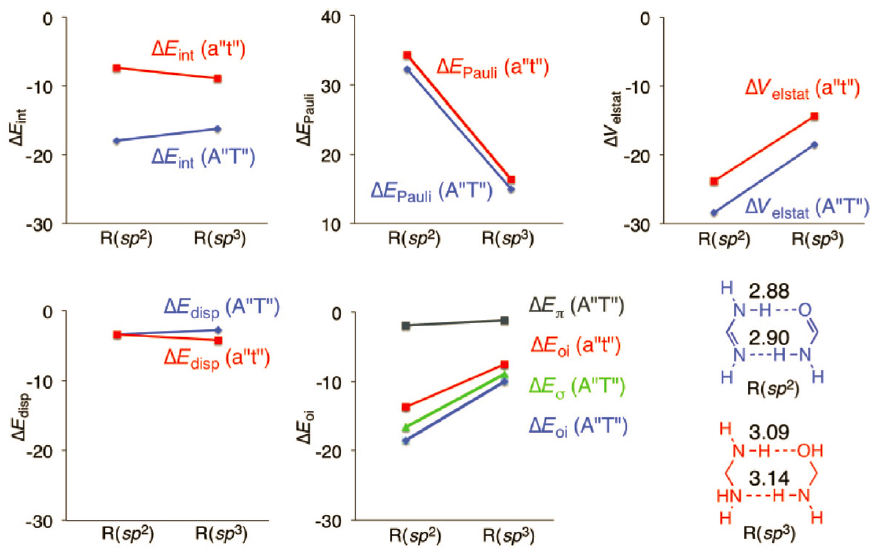
图9以图形形式展示了R(sp2)和R(sp3)距离的相互作用能分解。将A’’T’’和a’’t’’的距离从R(sp3)调整到R(sp2),增加的Pauli排斥必须要被键能的吸引作用部分抗衡掉(净剩余值为负数的相互作用能,否则就不是二聚体了)。静电相互作用,二者增加的量差不多,色散修正的改变不大。键长缩短给二者带来的最大改变是轨道相互作用:A’’T’’(△Eoi图中的蓝线)比a’’t’’ (△Eoi图中的红线)增加的更快。在该图中,分别显示了△Eoi的π和σ组分,可以看到主要是因为其中的σ组分(绿线)增加的更快所致。这导致A’’T’’的氢键平衡键长比a’’t’’短。
图9 R(sp)2和R(sp3)时,A’’T’’和a’’t’’的能量分解
现在我们的问题就变成了,为什么sp2杂化的σ组分的轨道相互作用比sp3更有利。为了理解这个问题,我们对A’’T’’和a’’t’’氢键在R(sp2)和R(sp3)时,Kohn-Sham分子轨道进行了分析(见表2)。表2中列出了前线轨道总的占据数和能量,以及前线轨道之间的重叠情况。A’’T’’中的N(H)…O氢键解释为0.05个电子从T’’的σHOMO转移到A’’的σLUMO和σLUMO+1(分别为0.01和0.02)。a’’t’’的电荷转移较小总共0.02电子从t’’的HOMO-1转移到LUMO和LUMO+1(各0.01电子)。二聚体的同一个氢键内电子施主与受主得失电子并不精确相等,因为还有极化导致的占据轨道与另一个单体的空轨道之间的混合。a’’t’’的电荷转移较小,从t’’的HOMO-1(-6.45eV),而T’’则是从σHOMO(-5.85eV)。A’’和a’’接受电子的轨道能级差不多大。A’’T’’和a’’t’’的N(H)…O氢键的前线轨道重叠大小也差不多。
A’’T’’的N…(H)N氢键的电子转移也比a’’t’’大。σHOMO贡献了0.09电子到T’’的接受电子轨道,而a’’的HOMO只贡献0.05电子到t’’的接受电子轨道。在这种情况下,HOMO-LUMO Gap不能用于解释这种差异,(A’’T’’和a’’t’’分别为5.2eV、4.8eV),HOMO-LUMO+1的Gap也不能(分别为6.0eV、5.7eV)。A’’T’’的轨道重叠几乎是a’’t’’的二倍,其中A’’T’’的 <σHOMO|σLUMO>为0.23,而a’’t’’的是0.10(表2)。这不过是因为A’’的σHOMO和T’’的σLUMO朝向正好配合的比a’’t’’更好而已(在Supporting Information图S3中可以看到)。
因此sp2杂化二聚体键长更短的原因归纳如下: 1)其中一个氢键(N(H)…O)的σ电子HOMO-LUMO Gap更小; 2)另一个氢键(N…(H)N)sp2杂化的前线轨道重叠比sp3杂化更好(即重叠值更大、重叠更多)
表2
ADF计算:
对于上面的表格,大多数数据的计算,前面都已经讲过了,剩下的就是计算单体(或者说片段)轨道的重叠情况,我们以A’’T’’的overlap为例说明:
1,优化A’’T’’的结构,在下载的计算文件中,对应的文件名字为:A2T2。需要注意的是,该结构初始构象已经是Cs群了,所以默认情况下(也就是在该任务的参数设置条件下),优化过程会自动保持Cs群。优化结束后,更新结构到ADFinput。
2,设置片段分析的参数如下图:
并如前面一样,将该二聚体分区,分区名字分别为A2、T2:
并确保片段功能启动:
默认情况下,程序不会输出SFO(片段轨道)的overlap,因此我们需要在图形界面上勾选一下:
如此,任务设置完毕,另存文件名为:A2T2-FragmentAnalysis(将另外产生A2T2-FragmentAnalysis.A2和A2T2-FragmentAnalysis.T2两个任务,总计新产生3个任务)。
3,在结果文件中,查找对应的数值:
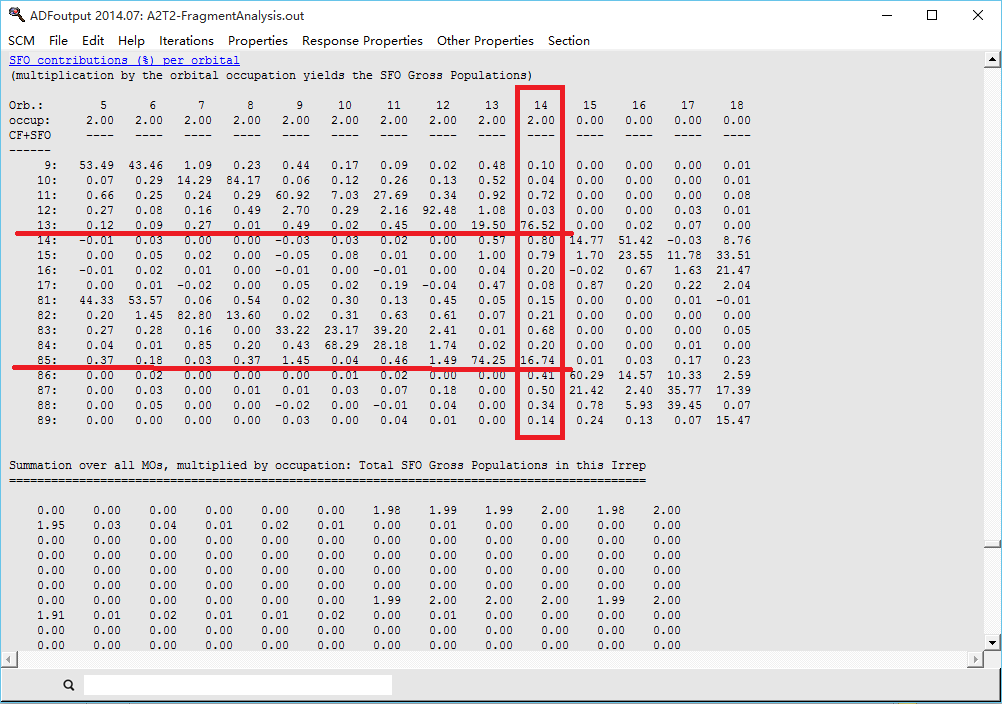
我们先要找到A’’的AA不可约表示(即σ)LUMO+1以及T’’的HOMO。在SCM LOGO > level中查看某个包含A’’的LUMO+1或T’’的HOMO二聚体轨道,例如14AA:
其轨道构成为来自A2的7AA和来自T2的7AA,而A2的7AA和T2的7AA都是各自单体的AA不可约表示HOMO。所以我们去out里面查看二聚体的14号轨道的构成列表(out窗口点击properties-SFO% per orbital):
注意SFO% per orbital有两个部分,分别为AA不可约表示和AAA不可约表示的。这里是AA不可约表示的,再往下翻,就是AAA不可约表示的SFO overlap和SFO% per orbital。
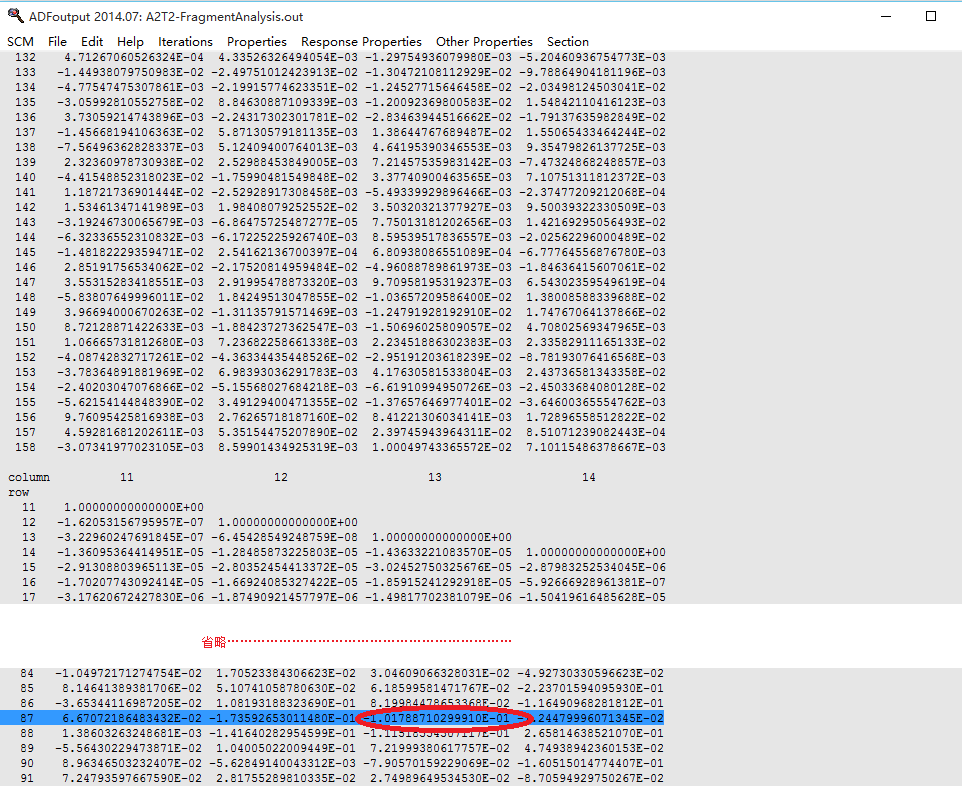
因此我们知道SFO里面编号13的就是T’’的HOMO,85就是A’’的HOMO,因此A’’的LUMO+1的编号就是87。那么<AσLUMO+1|TσHOMO>就是overlap矩阵的13行87列,或者87列13行。在out的该位置往上翻就能找到这个数据:
也就是上图红色圆圈的数据,是第87行、13列的数据:-0.10。文献中为0.11(正负号不重要)。
结论
本文研究了π对AT和同类模仿结构的协助作用。研究表明π的协助作用并不完全是因为芳香性;质子施主和受主原子的sp2杂化已经导致了π电荷的离域性。这个结论来自于对AT及其较小的类似模仿结构进行基于扩展性的,色散修正密度泛函计算分析。A’’T’’二聚体没有AT的芳香环,但氢键能与AT非常接近。A’’T’’以及其他AT的类似模仿结构在几何结构以及键能上的微小差别,通过定量的Kohn-Sham分子轨道和对应的能量分解分析(EDA)得到了解释。并揭示了π的协助作用与单体的π电子个数没有关系,但质子施主和受主必须拥有π电子,这样才能到达相近的氢键键能。
对sp2杂化的二聚体与sp3杂化的等价结构进行比较,并进行了Kohn-Sham分子轨道分析,如此而能理解导致氢键键长缩短、氢键增强的原因。A’’T’’(sp2杂化)的氢键能为-16.1kcal/mol,而a’’t’’(sp3杂化)的请见能为-8.2kcal/mol。这不能被解释为π电子的协助,因为π电子所带来的极化引起的能量仅为2kcal/mol。分子轨道分析揭示,sp2杂化的氢键更强是增强的静电相互作用和更好的共价相互作用所致。Sp2杂化氢键更短是由于两个原因所致:1) 其中一个氢键(N(H)…O)的σ电子HOMO-LUMO Gap更小;2) 另一个氢键(N…(H)N)sp2杂化的前线轨道重叠比sp3杂化更好(即重叠值更大、重叠更多)。因此,并非由于π电子的协助,而是由于不饱和二聚体与饱和二聚体相比,氢键的共价相互作用更强导致实验上我们法案共振协助(Resonance-assisted)AT及其较小的类似模仿结构氢键键长更短。