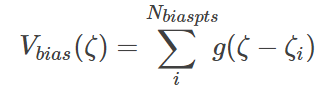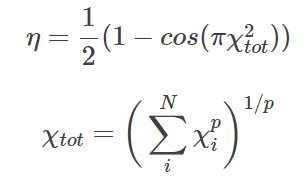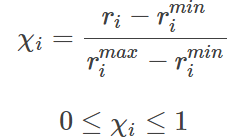目录
CVHD:加速分子动力学模拟中的裂解反应发生
Collective Variable-driven HyperDynamics (CVHD)原理
化学的动力学过程中,很多的重要问题,例如化学反应(催化、工业、酶)或物理过程(扩散、相变),都是由“罕见事件”驱动的,即发生该事件不太容易,以至于观察到该事件所需时间超出分子动力学可模拟时间范围之外(目前实际能模拟的时间尺度仅在纳秒级别)。
因此需要fbMC、REMD、Bond Boost等加速反应过程的方法,这些方法各有适合的范围,例如Bond Boost对于形成新键的反应非常适合,REMD对于有足够计算资源的情况下比较适合,fbMC往往在表面催化方面有很好的效果。而CVHD对于裂解反应,有很好的加速效果。
CVHD是一种“基于bias”的分子动力学,首先修改运动相关的势场/运动方程,以达到加速的效果(增大稀有事件发生的概率),然后从结果中去除“bias”的影响。
自由能法需要记录在每一步中添加的“bias”情况,需要定义合适的集合变量(CV)以降低这种方法的维数。一般而言,最多可以定义2~3个CV是可行(使用伞型采样、Metadynamics、自适应偏置力等方法),也可以使用NEB或String法沿一维反应路径取样。
无论是哪种形式,建立好的CV对结果非常重要,好的CV应该满足这些条件:
- 明确定义系统的状态(不同的状态不得交叠)
- 过程就平均而言是正交的
- 光滑、连续
因此CV通常不容易构造,不适合研究许多独立的同时过程。拿Metadynamics作为自由能采用方法为例来说明:bias通常为高斯函数,在当前CV值ξi处周期性地沉积。它逐渐填充能量值,直到没有剩余的能垒。
从而真实自由能就是填充完毕(即所谓“收敛”)之后填充函数Vbias的相反数,其中(g指Gaussian函数):
形象的说,一个坑的形状是什么?我们往里面倒入融蜡,之后取出蜡的形状取相反数,即坑的形状。
基于轨迹的“有偏采样”,可以作为自由能采样的替代方法,例如过渡路径采样或动态过程中的“实时无偏”,后者得到Hyperdynamics。“实时无偏”会丢失自由能信息,但时间可以从模拟中恢复。在修改后的势场上运行模拟时:
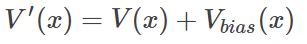
“hypertime clock”由下式确定:
简单地说,hypertime会告诉你“在无bias的模拟中需要多长时间才能到达这里?”。因此,为Hyperdynamics选择合适的bias(Vbias)困难且容易出错。
一种可能的补救方法是使用自学习bias,即CVHD。在这种方法中,使用Metadynamics及合适的CV η实时构建Vbias,但与Metadynamics相反的是,一旦检测到过渡(过渡态区域、反应发生),bias就会被重置,CV只需要对单个转换有效。
全局CV的η值由原始局部CV的χi构成(由最高χi控制):
此时只有某断键相关的局域CV生效 (参考文献:Bal and Neyts, J. Chem. Theory Comput. 2015):
Bias应用于接近过渡(态)时,通过键长rimax和rimin来定义过渡态特征(rimax值对应过渡态键长,rimin对应平衡态键长)。这样的CV构造,意味着当所有键在接近平衡态时,整体CV的η值接近于零。随着bias的增加,将η推向更高的值,这反过来又将系统中当前拉伸幅度最大的键进一步推向解离。一旦CV的η值达到1并在指定时间内保持该值,该算法即认为完成了过渡(反应),进而重置bias并更新键的情况。其中指数中的p是由用户设定的一个控制常数。
注意:
- 只有那些键级大于阈值(程序中需要预先设定的值,默认为0.5)的键才纳入CV的η计算范围
- AMS中各个计算引擎有自己的最小键级统计阈值,例如ReaxFF中键级小于0.3的键,将被统计为无键连,对此种情况,CVHD的阈值小于0.3就不合适
HyperDynamics与MetaDynamic的差别与联系
优势
HyperDynamics的优点:它是针对复杂系统的,目标是探索反应性,bias总是沿着一个特殊的CV设置,而CV是系统中总键数,因此系统倾向于断键。因为bias很具体,所以比MetaDynamic需要的用户输入参数要少。另一个优点是可以从中恢复出动力学信息,MetaDynamic则不能。
MetaDynamic的优点:如果要模拟单个反应,MetaDynamic通常效果最好。它能返回自由能表面,但没有动力学信息(至少不能直接得到动力学信息)。通过沿一组CV添加bias来加速模拟。在MetaDynamic中,CV必须由用户根据其对反应的了解来构造,因此MetaDynamic需要用户在这方面拥有专家级知识。
适用范围
- HyperDynamics:多个反应步骤,步骤未知
- MetaDynamic:单个反应步骤,用户是否大致了解其机制
AMS的PLUMED中也有MetaDynamic的接口,PLUMED有一个相当不错的CV库。CV是原子坐标的函数,PLUMED开发了一种语言来构造更复杂的CV。
下面是一个实例,在AMS分子动力学作业的MolecularDynamics字段中,添加了简单的PLUMED MolecularDynamics设置,用于展示脚本格式:
MolecularDynamics
InitialVelocities
Temperature 300
end
NSteps 1000
Plumed
Input
COORDINATION GROUPA=1 GROUPB=2-6 R_0=0.18 NN=6 LABEL=cv1
COORDINATION GROUPA=2 GROUPB=7-12 R_0=0.12 NN=6 LABEL=cv2
METAD ARG=cv1,cv2 SIGMA=0.02,0.02 HEIGHT=0.1 PACE=200 LABEL=restraint
PRINT ARG=cv1,cv2,restraint.bias STRIDE=1 FILE=COLVAR
end
end
Thermostat
Tau 10
Temperature 300
Type NHC
end
TimeStep 0.5
end
使用的CV是:
- 原子1和原子2~6之间的配位数
- 原子2和原子7~12之间的配位数
模拟将生成一个名为HILLS的文件,从中可以读取2D自由能曲面(需要AMS的脚本工具)。
本文主要介绍HyperDynamics的应用。
CVHD加速方法适用于AMS中ReaxFF、ADF、BAND、DFTB、MOPAC的分子动力学模拟
本文介绍基于ReaxFF的分子动力学中,如何调用CVHD达到加速热解反应的效果,在AMS中,也支持其他模块如BAND、DFTB、MOPAC的分子动力学调用该方法进行加速,除去各个计算引擎本身的方法参数设置之外,分子动力学参数、CVHD参数的设置与本文完全一致(图像界面是一致的)。
本文选取了一个简单的例子:模拟实际条件下(1000k,50kg/m3)十二烷的低温热解。这是一个具有挑战性的模拟反应,因为烷烃的热解产物取决于温度,导致以下趋势:
- 高温(>2000K)- 乙烯是主要反应产物,因为熵优势分解反应成为主要过程
- 较低温度 - 更大的1-烷烃的形成,导致主要形成较大的产物分子C3,乃至更大的产物。
因此,不能仅仅通过提高模拟温度来模拟低温热解的动力学。此外,考虑到键断裂反应的罕见事件性质,依靠延长动力学模拟时间的蛮力方法是不可行的,如下文所示,十二烷断裂发生的时间尺度在毫秒级。
本文模拟结果参考文献:
- Kristof M. Bal and Erik C. Neyts, Direct observation of realistic-temperature fuel combustion mechanisms in atomistic simulations, Chem. Sci., 2016, 7, 5280
与一般的反应分子动力学模拟不同的是,CVHD要求步长更小(例如0.1fs)从而精度更高,本文为了更快得到结果,使用了0.2fs步长,体系仅包含几百原子。因此可以估计各种反应发生的时间尺度,但诸如速率常数等则因为样本数太少而缺乏统计平均的意义。
这种加速方法,适用于中等规模的体系:CV相关的键的数量最好在1000个左右,或者更少一些,当然略多一些问题也不大,数量太大CVHD的加速效果就不明显了,因为会太频繁地触发事件,从而限制bias累积速度,进而限制裂解加速效果。
ChemTrazYer2.0支持基于CVHD模拟的结果的基元反应分析。
分子动力学参数设置
模型
一般性参数设置
选择力场CHO(不同模块,例如ADF、BAND、DFTB、MOPAC,分子动力学参数设置区别仅在此窗口,后面的分子动力学参数设置、CVHD参数设置与本教程所示完全一致):
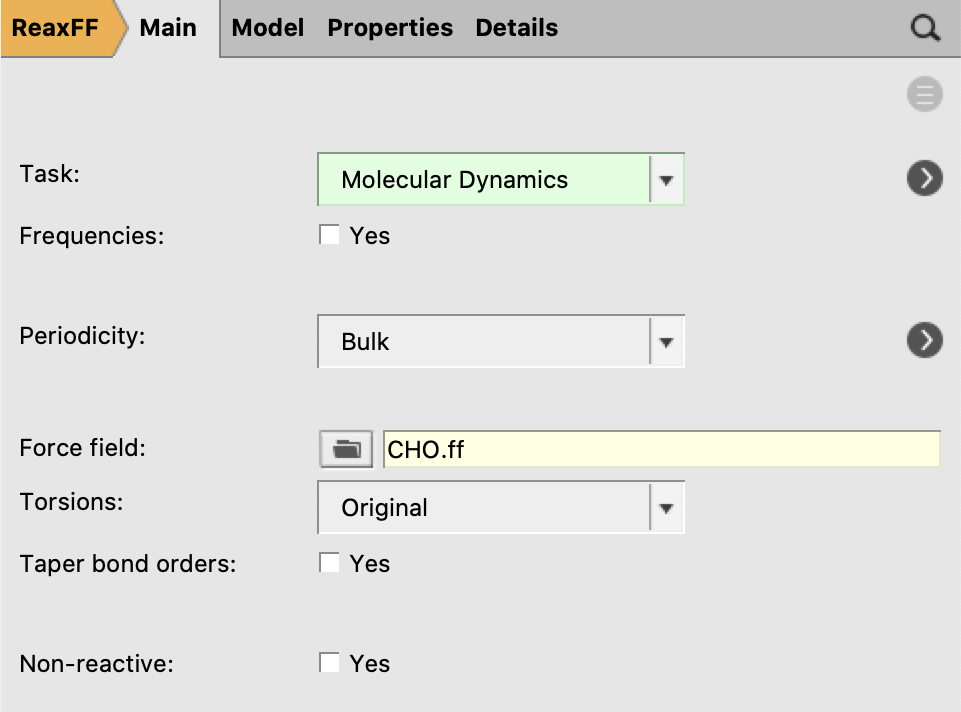
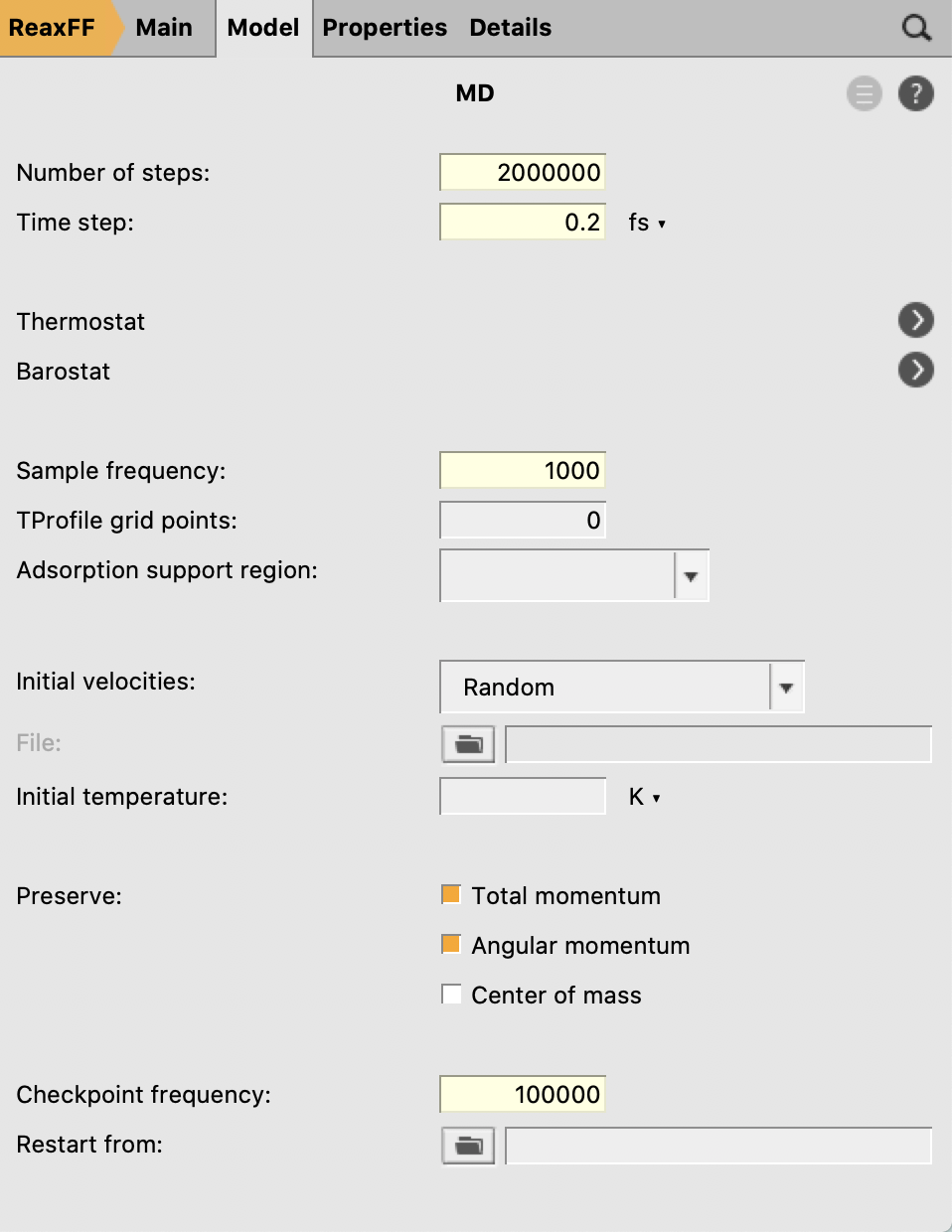
设置总步数、步长(如上所述比一般MD偏小,这里设置为0.2已经偏大,建议设置为0.1)、轨迹文件保存频率(如果不关心轨迹本身,则该值可以设置很大例如1000,如果关心轨迹本身,例如需要通过轨迹观察反应的路径,则需要设置的较小例如100)、checkpoint保存频率一般都设置的比较大,例如10万步:
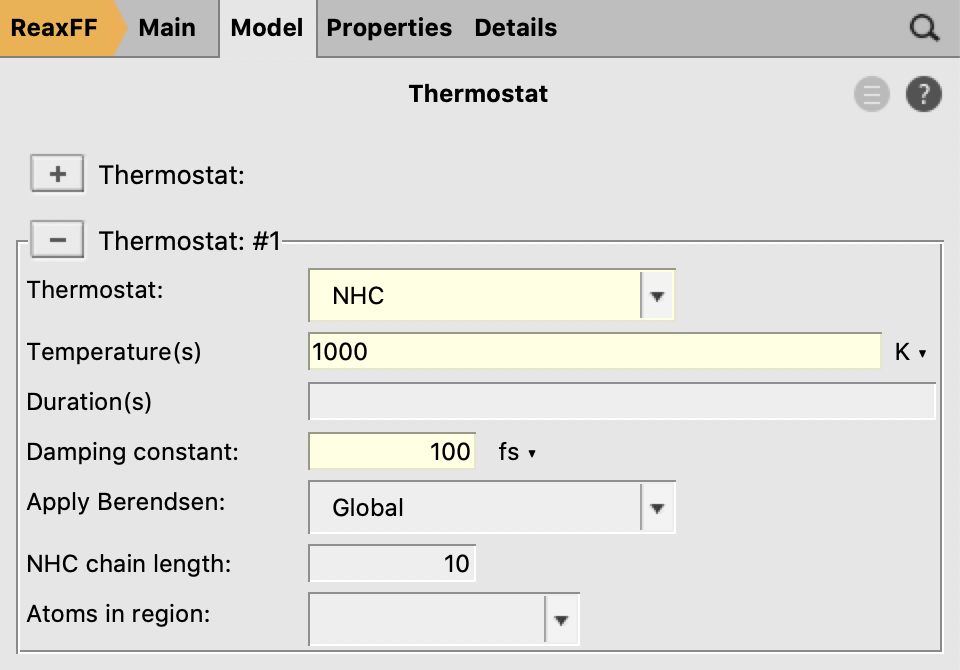
设置温度为实验温度1000K,Damping Constant必须设置大于0的值(100fs以内),但一般对结果没有太大影响:
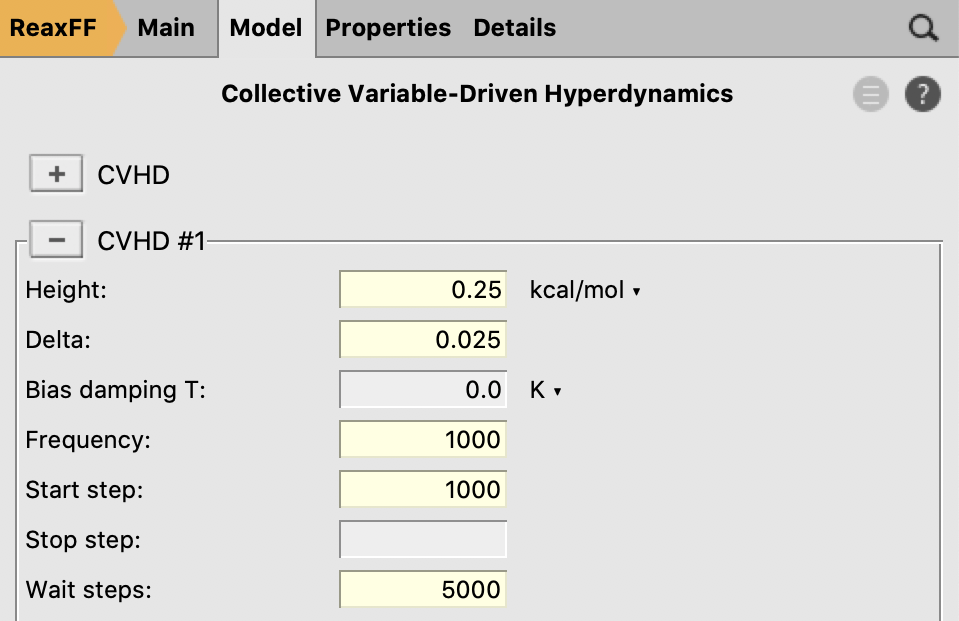
CVHD参数
这里是热解反应,因此涉及到C-C、C-H键的断裂,因此设置两个局域CV:
- C-C: Rmin = 1.55 Å , Rmax = 2.10 Å
- C-H: Rmin = 1.05 Å , Rmax = 1.65 Å.
注意最小值对应平衡键长,最大值对应过渡态键长。设置如下:
Model → Collective Variable-Driven Hyperdynamics
- Height:添加的bias(是一个Gaussian函数)的高度,该值需要根据预估反应能垒的高低,略微调高或调低,从而调整bias沉积速率
- delta:Gaussian函数的宽度
- Bias Damping T:指添加Bias的时候,是否使用衰减方式(衰减趋势为exp(-E/kT)),即接近顶部时,Gaussian函数有所减小,T为0则不启用衰减
- Frequency:添加新bias Gaussian函数的频率,以步长为单位。如果当前CV的η值小于0.9,步数大于或等于Start Step,小于或等于Stop Step,则每隔Frequency这么多步,沉积一次新的bias Gaussian函数。该值需要根据预估反应能垒的高低,略微调大或调小,从而调整bias沉积速率
- Wait Step:如果CV的η值变为1,并且在这么多步中保持该值,则认为反应事件已经发生。在此之后,将重置CV,并消除bias
点击Collective Variables前面的➕两次(对应两个CV),并增加如下参数设置:
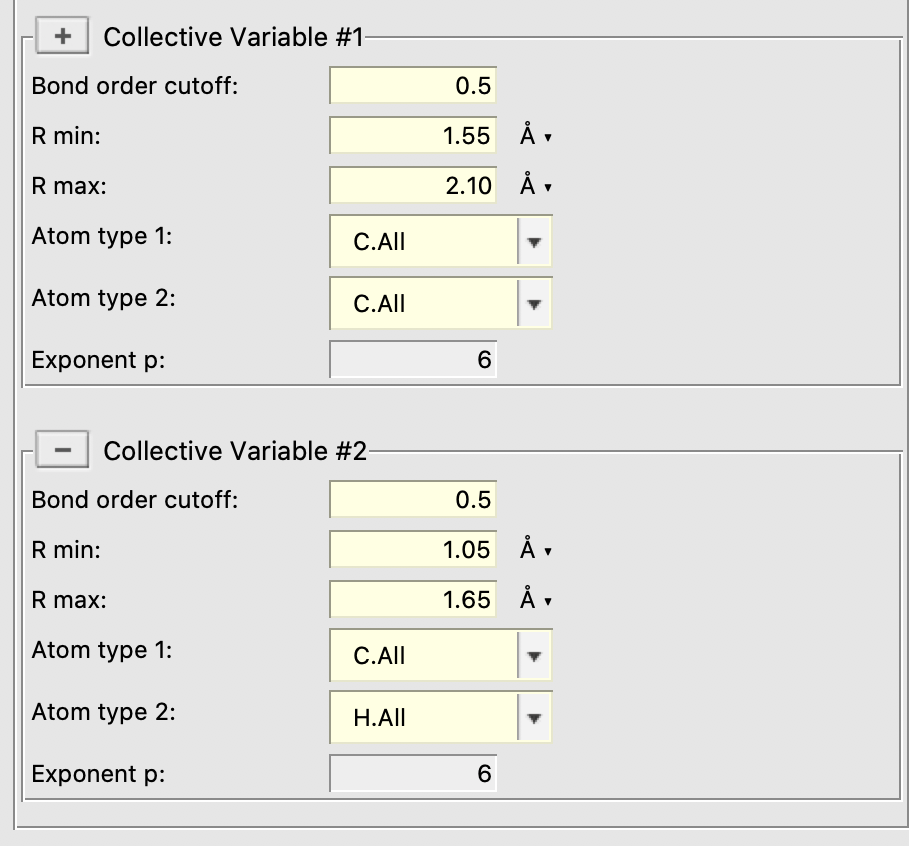 其中
其中
- bond order cut off即上文说的阈值,低于该阈值的键级不纳入CV的η值计算范围,一般默认0.5即可
- Rmin与Rmax含义如上文所述
- Exponent P含义如上文中CV的η值相关公式所示
保存作业并运行。
结果查看
如果不启动CVHD,其他参数与条件保持不变,则无法观察到任何反应。但启用的情况下,发生了多次裂解反应。
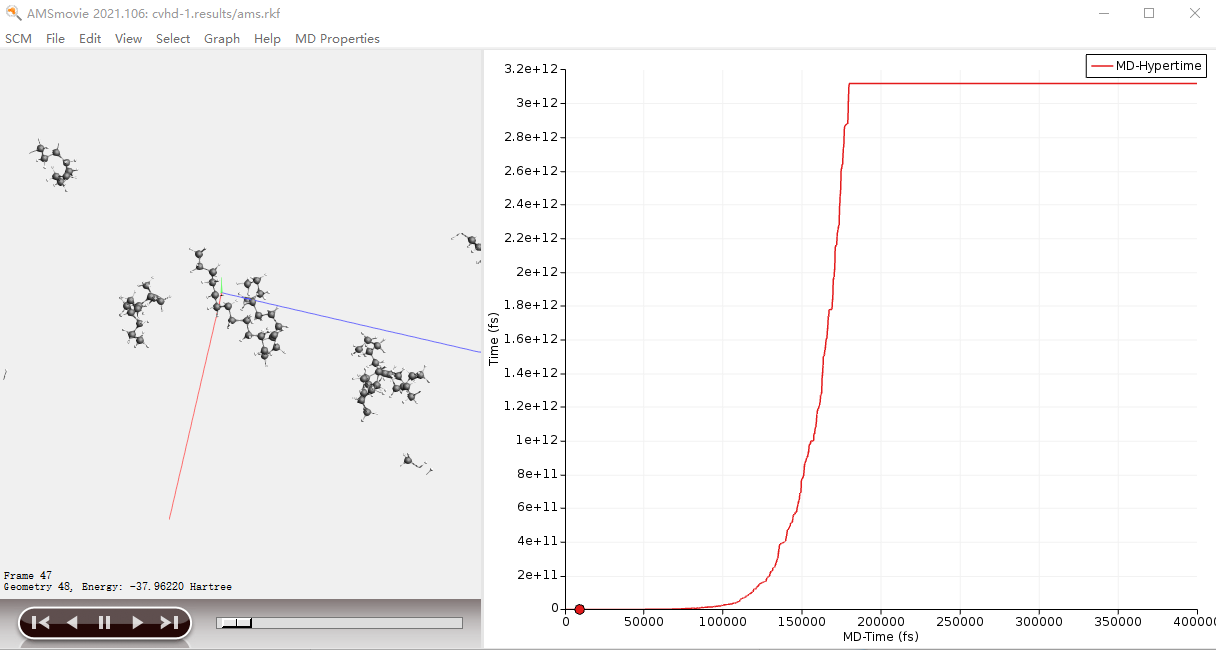
HyperTime
如前面所述,HyperTime是无bias时的“真实”时间尺度,即在没有CVHD的情况下一个进程需要多长时间。SCM - Movie - Graph - delete Graph去掉默认显示的能量变化曲线,然后MD Properties - HyperTime,可以看到纵坐标为HyperTime,横坐标为帧。可以把横坐标改为MD时间:MD Properties - time - Graph - Curve on X,则x轴变为了MD的时间。
双击纵坐标,也可以修改y轴的单位:
组分分析
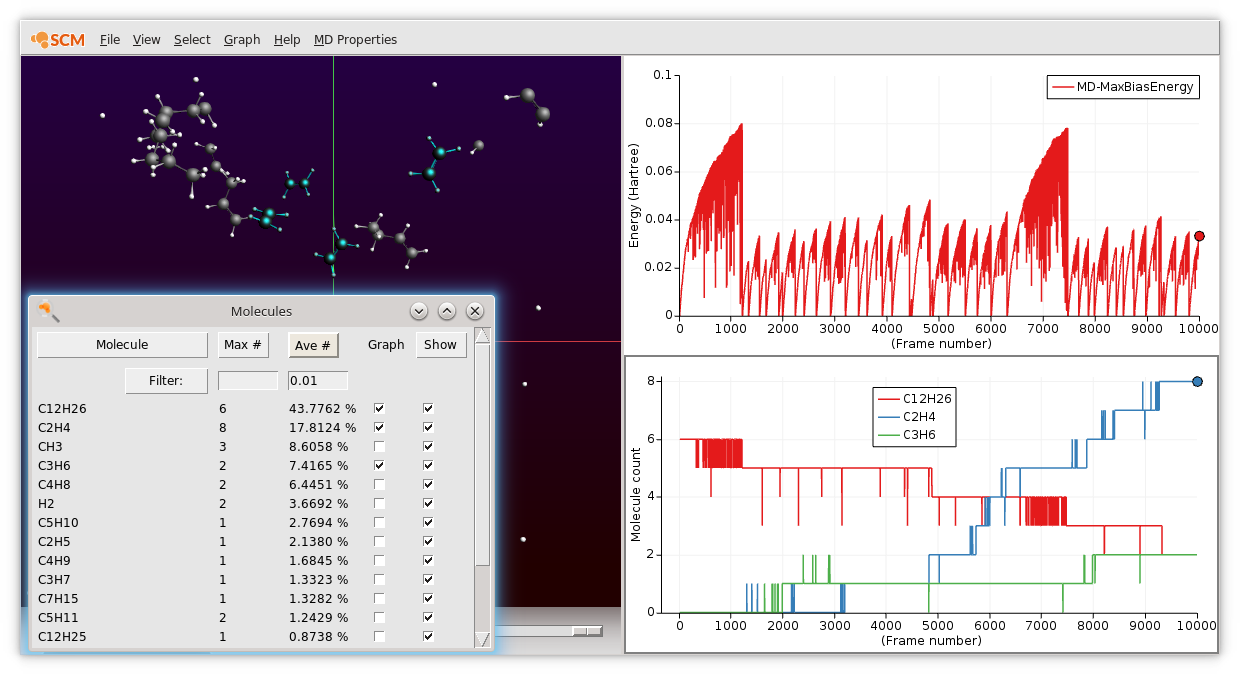
在分子动力学模拟运行过程中,您可以使用AMSmovie检查模拟过程。例如,可以通过Max Bias Energy图,查看CVHD的bias是如何逐渐形成的,以及事件发生时如何的重置:
delet键删除之前的Graph,然后
- MD Properties → Max Bias Energy
- Graph → Add Graph添加一个新的曲线窗口,在该窗口显示产物变化曲线:
- MD Properties → Molecules
可以看到一个反应有效发生的时候,就存在一次Max Bias Energy的突变——清零。
bias沉积图
用户可以在命令行,用Python脚本生成。命令行打开方式:
- 对Windows来说,在命令行执行的具体方式为:执行AMS202*.*\ams_command_line.bat,将自动读取环境变量。输入sh回车,之后即可像Linux一样,进行命令行工作
用户输入命令:
$AMSBIN/amspython $AMSHOME/scripting/standalone/reaxff-ams/cvhd-hills.py */*.results回车
注意:
- 第一个$符号不要遗漏
- */*.results替换为作业生成的results文件夹的完整路径
- 可以复制上述内容,在命令行粘贴后修改*号部分即可
回车后将生成一个名为cvhd-hills.csv的文件,并由AMSgraphs自动打开。修改显示方式(不要以曲线的方式,而改为以分散点的方式显示):Plot → Options → Curves,去掉Curve的勾选,并勾选Data Points:
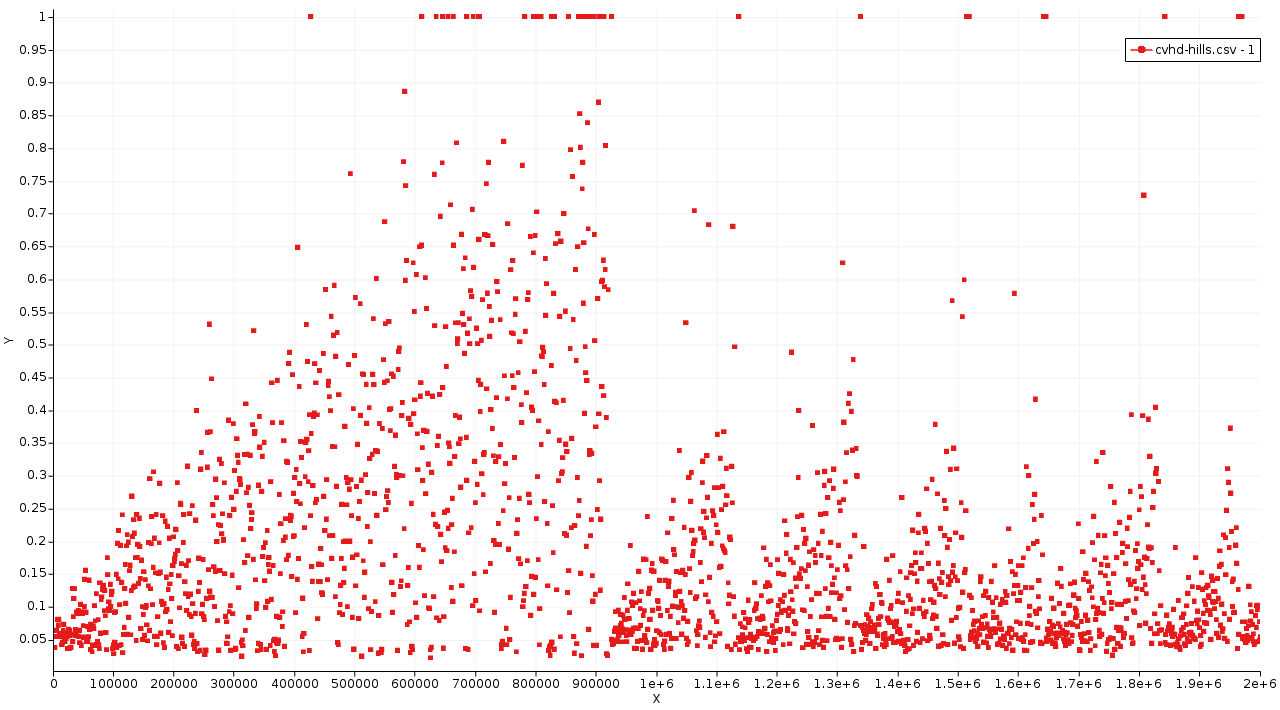
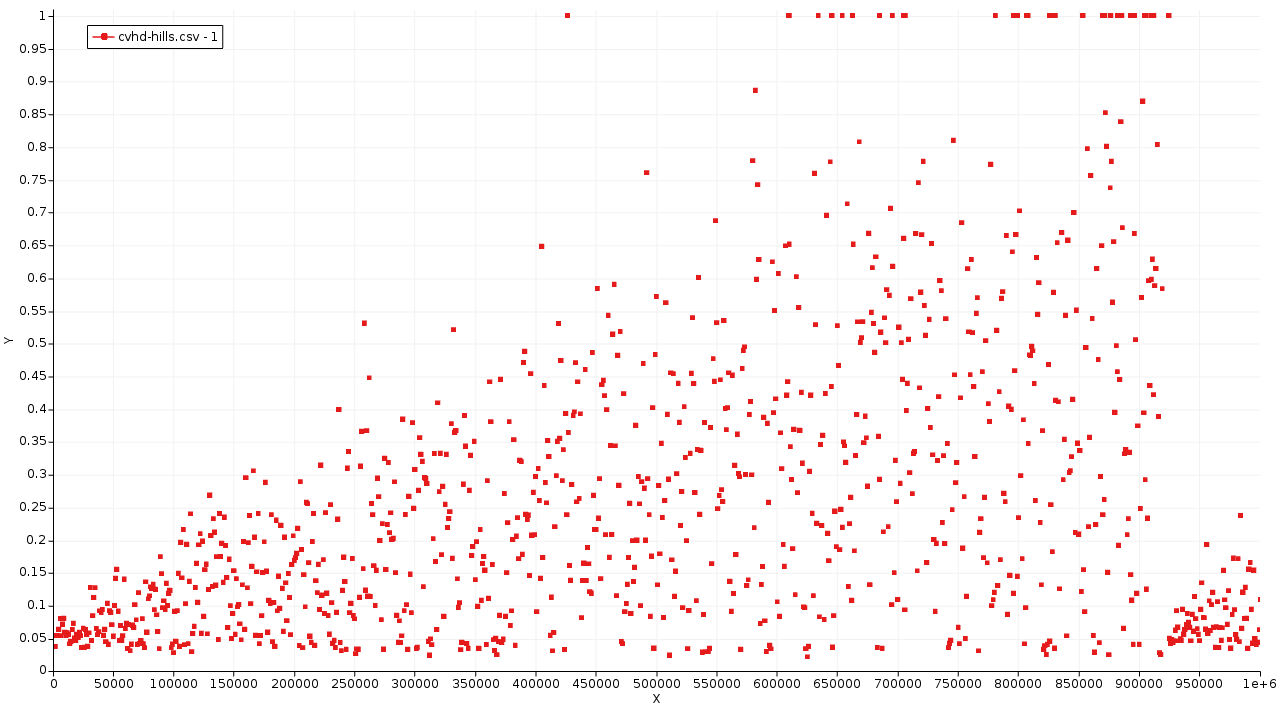
图中横坐标是Step,纵坐标是CVHD的全局CV的η值。每个点代表一个的Gaussian bias峰,但η=1处的点仅表示系统访问了过渡区,CVHD方法不会在η>0.9的区域有bias。
为了更深入的理解CVHD,我们可以详细看看上图中,第一次反应事件的部分:
最初,系统保持在η=0左右(所有键处于平衡值附近)。随着偏压的增大,η逐渐被推到越来越高的值(键被拉伸得越来越长),在42500步左右η达到1.0,但随即返回到较低的值。这意味着在指定的等待时间内没有完成过渡(即没有发生反应)。最后,约90万步左右,η在1.0处停留了足够长的时间(对应我们在参数设置里面的Wait Steps),表明键已解离。
对上图的跟踪观察,有助于诊断、改善CV的设置:
- 初始η不接近零:Rmin设置得太低。CVHD“认为”平衡结构已经部分解离了
- η不能自由够到更高的值(长时间保持接近零的值):Rmin设置得太高
- 太多的recrossings(η继续达到1.0,但没有发生真正的反应):Rmax设置得太低,远小于过渡键长
- 系统从低η直接跳到1.0,触发反应:Rmax设置得太高,超过过渡态键长
每个局域CV单独测试调整,然后组合一个模拟作业中,通常会很有帮助。全局CV包含局域CV的加权和,从而产生一个最佳Rmin,一般,体系越大,Rmin也需要设置的略大。
分析反应时间的时间尺度
同样地,需要在命令行使用Python脚本生成一个时间图:
$AMSBIN/amspython $AMSHOME/scripting/standalone/reaxff-ams/cvhd-hypertime.py */*.results
回车后,AMSGraph将自动打开时间图,我们可以将图的y轴更做对数,从而显示比例会更好看一些:
- Plot → Options → Left Y axis,确保Y轴最小值是一个正数,例如1e-12
- 勾选Logscale
- Plot → Options → Curves
- 去掉Curve的勾选,并勾选Data Points
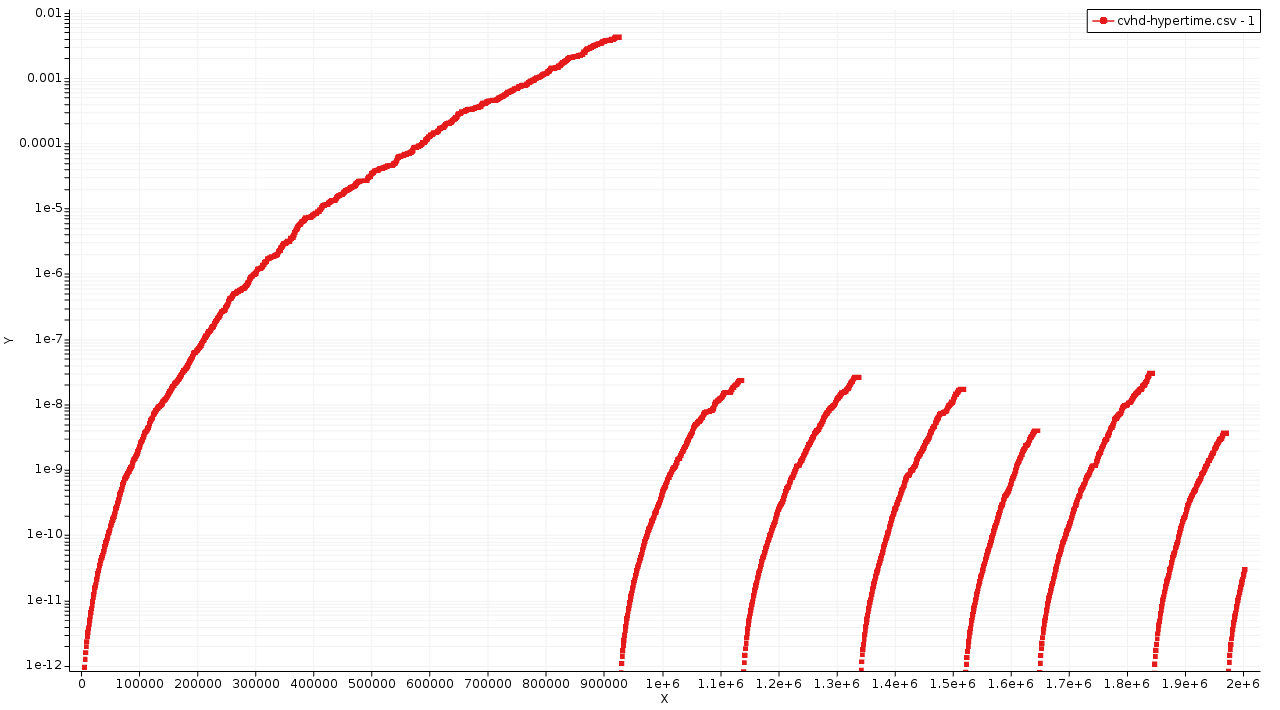
x轴显示Step,y轴显示自上次事件以来的HyperTime(以秒为单位)。每条曲线都显示了随着bias的加入,时间逐渐加速,直到检测到事件(键分离)。该图中,不同时间尺度,可能对应了不同类别的反应过程:
- 第一个过程(十二烷链断裂)时间尺度在毫秒级别
- 后续步骤发生的时间尺度在ns~μs级别
该曲线一般而言比较平滑,否则CVHD的参数(Rmin、Rmax、Frequency等)设置可能需要改进。
讨论
为了提高计算效率,本教程使用了一个小示例系统以及最少的步骤。直接后果:
- 我们不能期望任何合理的统计数据或速率常数
- 大多数基本反应只观察到一次,有些完全没有观察到
- 只能对时间尺度进行粗略的数量级估计
- 多次模拟,可能产生大不相同的结果
为了克服这些限制,需要更大的系统和更长的模拟时间。这里选择的时间步长0.2 fs,对过渡期间而言可能导致结果不准确,可能使用较短的时间步长(0.1 fs)更好。本例中的C-H键的Rmin/Rmax参数,是通过检查bias沉积图,进一步调整得到的。
本文参考SCM官网英文教程:https://www.scm.com/doc/Tutorials/MolecularDynamicsAndMonteCarlo/CVHD.html