2016以前的版本如何进行巨正则系综蒙特卡洛GCMC模拟
体系与一温度为T的粒子(分子、原子)源接触,体系不仅同粒子源有能量交换,而且可以同粒子源有粒子的交换,最后达到平衡,这种系综称巨正则系综,可以用于模拟块体表面吸附小分子、原子的反应过程。
以铁表面的吸附O2、H2O为例来说明。
建模:
在美国矿物学家晶体结构数据库可以下载Fe晶体:
ADFinput > File > Import Coordinates:
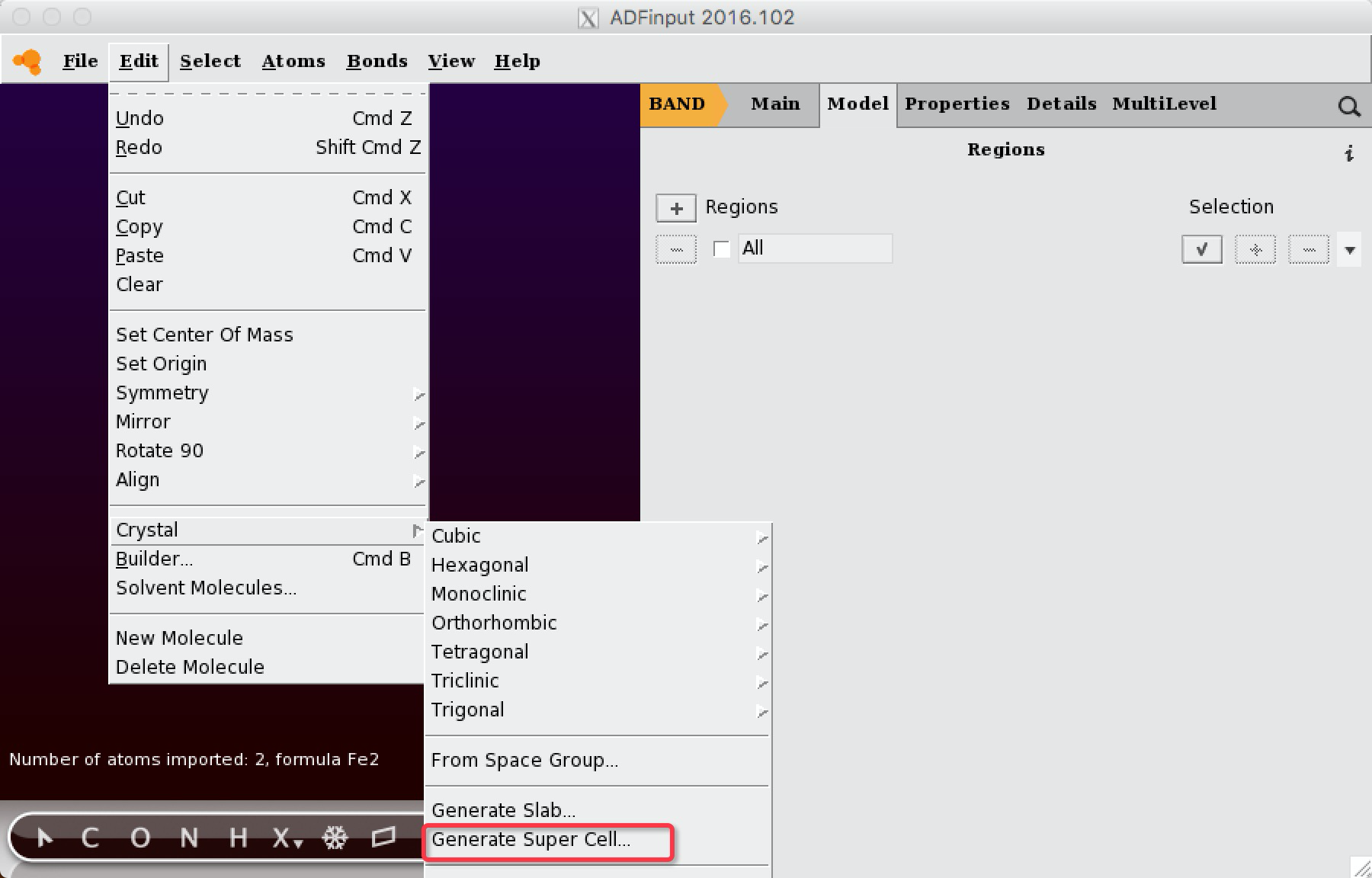
之后点击右下角的四个圆点图标,将默认的3*3*3周期显示切换成只显示一个周期:
创建5*5*2的超包:
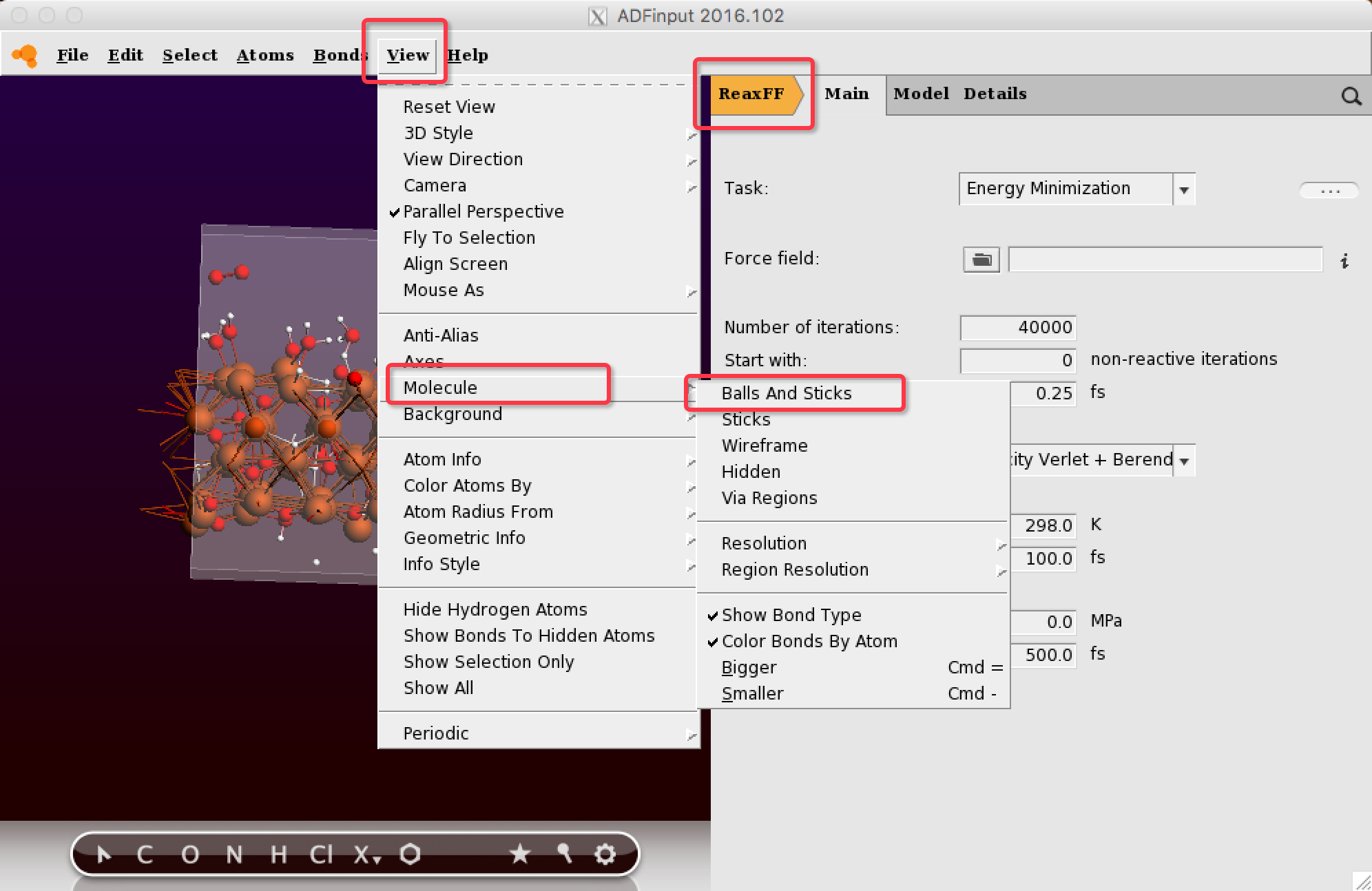
切换到ReaxFF模块:
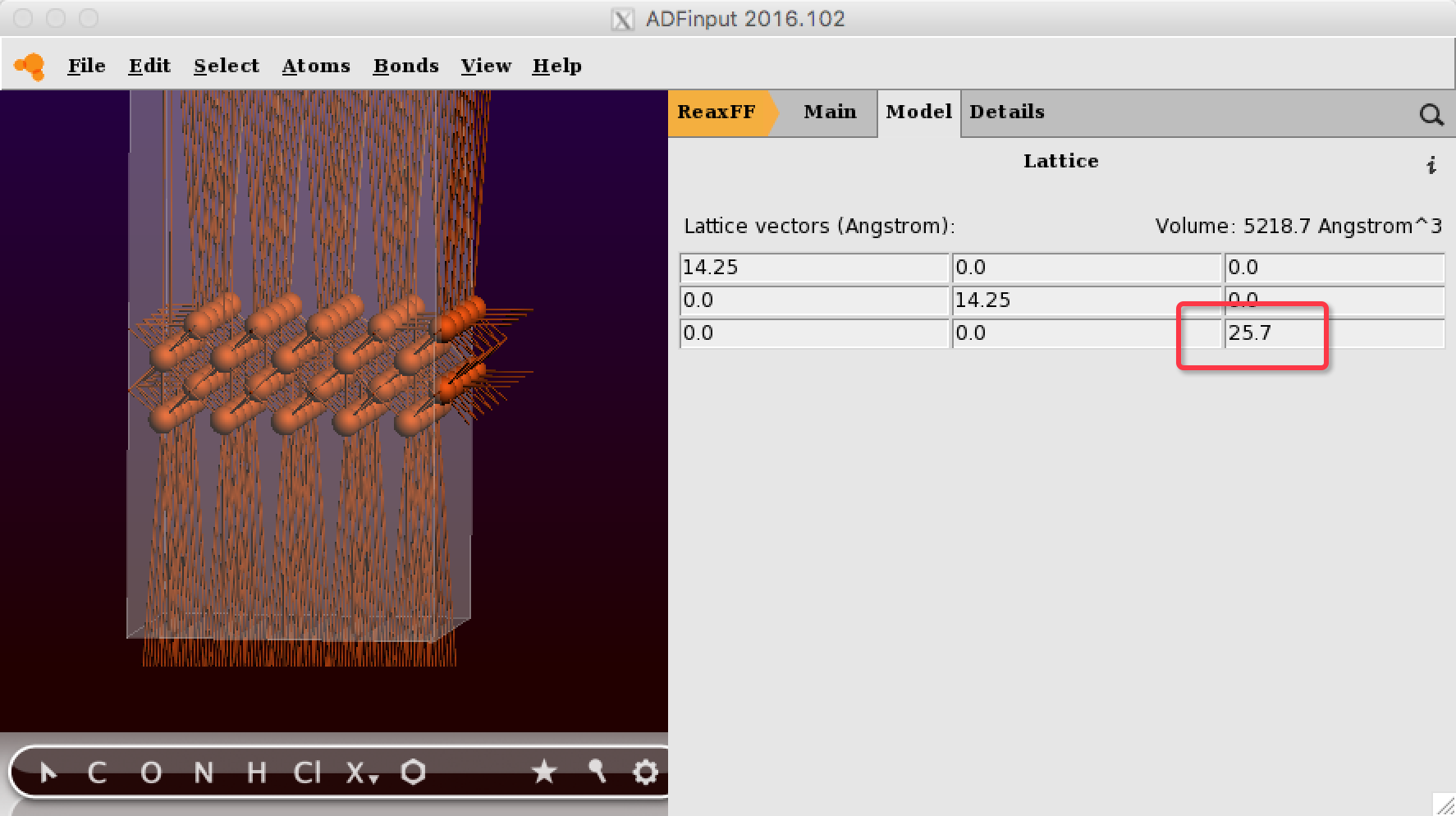
之后,在Model > Lattice修改晶格常数,将C方向,酌情增加一层真空,例如这里原先厚度为5.7埃,我们增加20埃到真空,C方向的Cell尺寸增加20埃。
之后,可以看到Bond的显示有些问题,有的键被拉的很长——这本身不影响模拟结果,但不美观,因此我们点击菜单Bonds > Removie Bonds这样,去掉所有的Bond的显示,之后Bonds > Guess Bonds,重新生成Bond的信息。
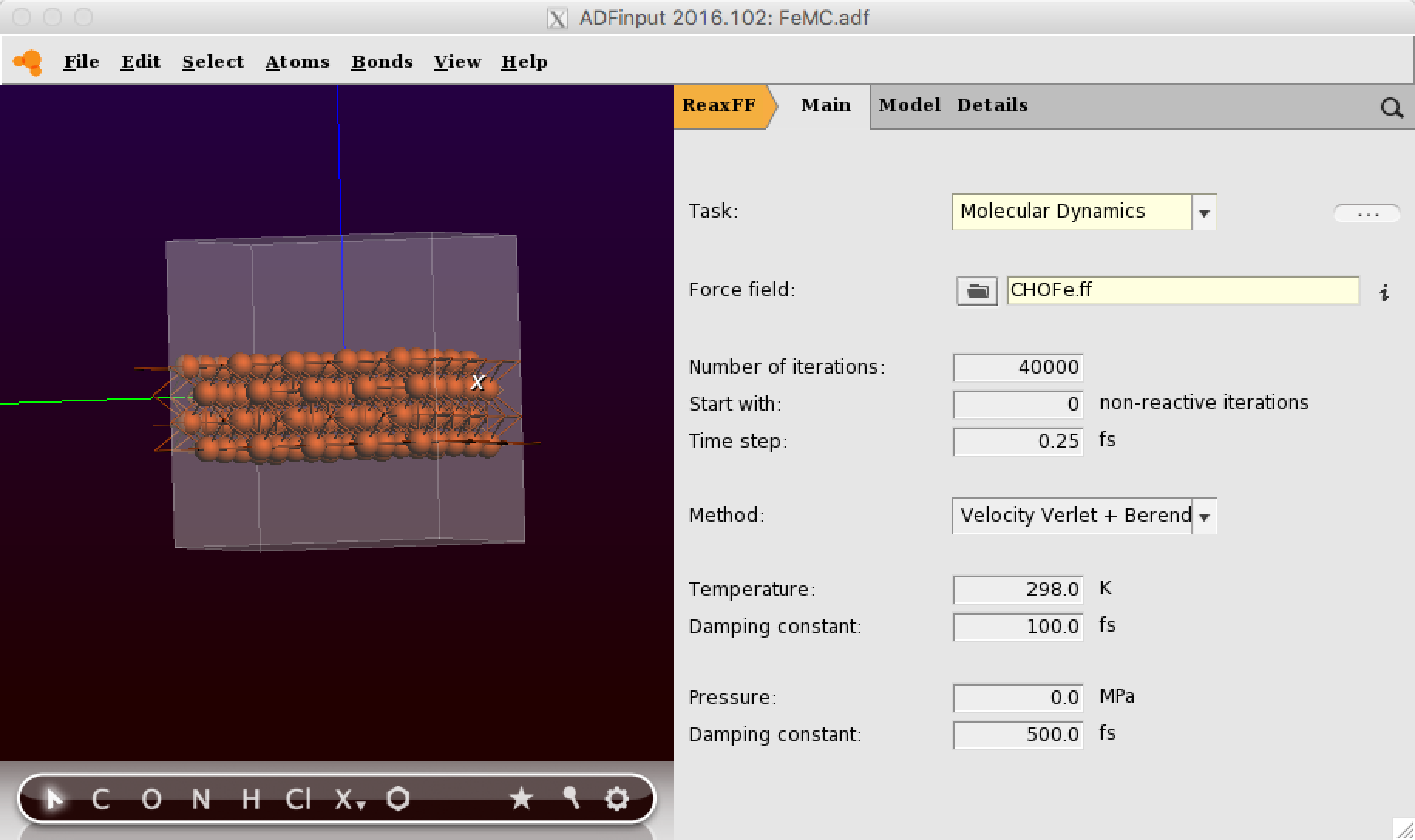
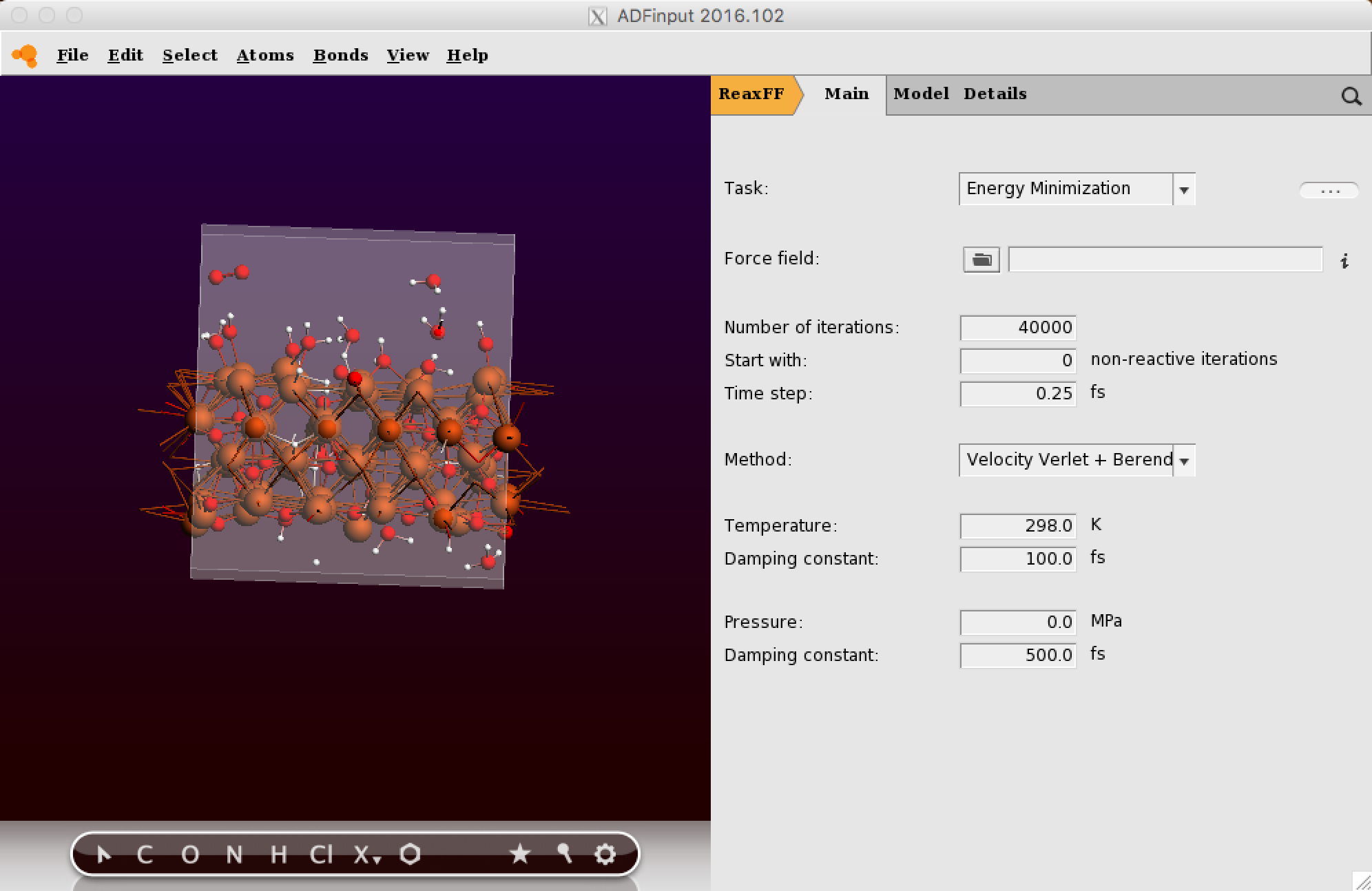
设置基本的模拟参数,包括力场等:
添加小分子:
在表面上方一段距离(例如2埃以外)创建O2、H2O分子各一个,之后保存。
设置GCMC参数
打开*.run文件,做如下修改:
注意:拷贝下面的这些脚本的过程中,需要留意粘贴过去之后,格式是不是变化了,包括:空格数是不是不一样了,小数点的对齐方式是不是不一样了?上面的下载文件中的*.run文件是经过运行验证的,因此可以作为很好的参考。
1,删掉
cat > control <<eor …… eor
替换成:
cat > control <<eor # some of the parameters that influence the minimization step in the GCMC code 1 icentr Put the center of mass at the center of the cube 1 igeofo 0:xyz-input geometry 1: Biograf input geometry 2: xmol-input geometry 2.50000 endmm End point criterium for MM energy minimisation 500 imaxit Maximum number of iterations 0 icelop Optimize cell parameters 0=no 1=yes 1.00050 celopt Cell parameter change 0 imaxmo In this case: 0: POLAK_RIBIERE Conj.Grad method, 1: Limited-memory BFGS method eor
2,在上面的内容后面,添加如下内容:
cat > iopt <<eor 5 eor
以及添加如下内容:
cat > control_MC <<eor ! GCMC control file example 0 iensmb !select MC ensemble (0=Mu-NVT with fixed volume, 1=Mu-NPT with variable volume) 5000 niter !number of MC iterations to do this simulation 0 nstart !start the iteration counter with an offset, usefull for restarts to avoid double files 300.0 mctemp !Temperature of the simulation, affects acceptance rate for steps that increase the energy 0.0 mcpres !NPT pressure in GPa (set to zero for incompressible solid systems unless at very high pressures) 3.0 rmaxpl !Max radius for atom placement on insert/displace move 1.2 rminpl !Min radius for atom placement on insert/displace move 2000 nmctry !Maximum number of trials allowed when inserting or moving a molecule. If the ! ! rmaxpl and rminpl variables are very strict, this number needs to be large 1 igcfac !include GC prefactor in probabilities? 0 = no 1 = yes 0 ivol !select MC volume calculation technique: ! ! 0: vvacu needed! volume = total volume - occupied volume - specified vacuum volume (vvacu) ! ! 1: volume = total cell volume ! ! 2: vacc needed! volume = specified accessible volume (vacc) ! ! 3: volume = total cell volume - occupied volume ! ! 4: vacc needed! volume = specified accessible volume (vacc) - occupied volume 435.0 vacc !if ivol=2 or ivol=4, specify Vacc in angrstoms^3 0.0 vvacu !if ivol=0 specify non-accessible (vacuum) volume Vvacu in angrstoms^3 0.25 ivlim !volume change limit (value between between 0 and 1, Vnew = ((1+ivlim)*V1) 1 resopt !write restart files: 0=no, 1=yes 1 resfrq !frequency of writing restart files (MC code only writes files if the MC move is accepted) 0 debug !print debug output if set to 1, print even more debug output when set to 2 1 nmols !Number of MC molecule types, must match the number of molecule blocks that follow! ! !Molecule Specific Data: H2O Example -75.00 cmpot !chemical potential of molecule 1 noinsr !setting this to 1 disables insert/deletion moves. If it is set to 1 for all types, the ensamble becomes NVT/NPT 3 nmatom !number of atoms in molecule -4.9071400000000000 1.804740000000000 4.595350000000000 O -5.2042200000000000 2.419330000000000 3.920340000000000 H -4.0159200000000000 1.511840000000000 4.391540000000000 H !Molecule Specific Data: H2 Example -75.00 cmpot !chemical potential of molecule 1 noinsr 2 nmatom !number of atoms in molecule 3.426300000000000 -1.2337700000000000 3.477750000000000 O 2.147280000000000 -0.7508700000000000 4.070210000000000 O eor
上面的脚本中,最后面的12行内容是关于H2O、O2的坐标。该坐标是从*.run文件的前面部分“剪切”(也就是*.run脚本中不再有H2O、O2的坐标)过来的。*.run中坐标的列表类似如下:
HETATM 91 Fe -2.85000 5.70000 0.00000 Fe 1 1 0.0 HETATM 92 Fe -1.42500 7.12500 1.42500 Fe 1 1 0.0 HETATM 93 Fe -2.85000 -5.70000 -2.85000 Fe 1 1 0.0 HETATM 94 Fe -1.42500 -4.27500 -1.42500 Fe 1 1 0.0 HETATM 95 Fe -2.85000 -5.70000 0.00000 Fe 1 1 0.0 HETATM 96 Fe -1.42500 -4.27500 1.42500 Fe 1 1 0.0 HETATM 97 Fe -2.85000 -2.85000 -2.85000 Fe 1 1 0.0 HETATM 98 Fe -1.42500 -1.42500 -1.42500 Fe 1 1 0.0 HETATM 99 Fe -2.85000 -2.85000 0.00000 Fe 1 1 0.0 HETATM 100 Fe -1.42500 -1.42500 1.42500 Fe 1 1 0.0
注意事项
注意1:
脚本中,H2O、O2的坐标格式要求非常严格。最好是参考https://www.scm.com/doc/ReaxFF/GCMC.html
将其脚本内容直接拷贝过来,然后一个一个原子坐标替换——尤其是坐标的数字的位数一定要与该文中一致,否则会报错。
上面添加的其他控制脚本,最好也都从https://www.scm.com/doc/ReaxFF/GCMC.html直接复制过来(因为WIKI页面语法的缘故,格式有些变形了)。脚本修改好之后,保存。
注意2:化学势的计算
关于‘cmpot !chemical potential of molecule’这一项,上面的例子中,化学势都设置为-75kcal/mol。化学势计算公式: [μ(T,P)+kbT*ln(P/P')-Ed]/2
- μ(T,P)是在温度、压强下,实验测得的化学势
- kb为波尔兹曼常数,T为温度
- P'是小分子的分压强。如果只有一种气体,那么kbT*ln(P/P')=kbT*ln1=0
- Ed是分子的解离能