目录
如何为 QuantumATK 创建新的附加组件
在 QuantumATK,附加组件是包含一个或多个插件的 Python 模块,可用于向软件添加新功能。有几种类型的插件可供选择。本教程将关注允许 QuantumATK 读写新数据格式的插件类型。在本教程中包含了三个示例。第一个是从 XYZ 文件中读取分子构型的插件,第二个是读取电子密度的插件。 ^  ^
^
附加组件模块的基本结构
因为插件是包含在 Python 模块中的,所以它们应存在于自己的目录中。我们将研究一个读取 XYZ 文件的插件作为示例。它的目录结构如下:
XYZFilters/
__init__.py
XYZLabFloor.py
XYZFileRawReader.py
init.py 文件是一个特殊文件,用于告诉 Python 该文件夹是一个模块。它还包含导入模块时执行的代码。对于插件,此文件还需要包含一些信息用用以告知 QuantumATK 关于它自身。
例一:读取 XYZ 文件的插件
XYZFilters 附加组件中的 init.py 文件包含以下代码:
1 import datetime 2 import XYZLabFloor 3 4 __addon_description__ = "Plugins for importing and exporting XYZ configurations." 5 __addon_version__ = "1.0" 6 __addon_date__ = datetime.date(year=2015, month=8, day=6) 7 8 __plugins__ = [XYZLabFloor.XYZLabFloor] 9 10 def reloadPlugins(): 11 reload(XYZLabFloor)
此代码给出了附加组件的说明,分配版本号和上次更新的日期。它还定义了附加组件提供的插件列表。本例中,有一个 XYZLabFloor.py 文件,包含了一个名为 XYZLabFloor 的插件类。此外,还提供了一个名为 reloadPlugins 的函数,以便在源代码文件发生更改时,QuantumATK 可以重新加载插件。
我们现在来看一下包含插件类的 XYZLabFloor.py 文件的结构。在文件的顶部,我们导入了编写插件所需的模块和类:
1 import os 2 3 from API import LabFloorImporterPlugin 4 from API import LabFloorItem 5 from NL.CommonConcepts.Configurations.MoleculeConfiguration import MoleculeConfiguration 6 from NL.CommonConcepts.PeriodicTable import SYMBOL_TO_ELEMENT 7 from NL.CommonConcepts.PhysicalQuantity import Angstrom 8 from NL.ComputerScienceUtilities import Exceptions 9 10 from XYZFileRawReader import XYZFileRawReader
接下来我们需要定义插件类。插件必须从 QuantumATK 定义的特定类中继承。在这种情况下,我们将定义一个从 LabFloorImporterPlugin 继承的类:
13 class XYZLabFloor(LabFloorImporterPlugin):
这种类型的插件必须定义两种方式。第一种方式是 scan。该方法的作用是确定插件是否处理特定文件,如果是的话,则确定该文件包含哪种类型的 LabFloor 对象。它会将项目列表返回给 LabFloor。第二种方式是 load,负责解析文件并将对象加载到内存中。
对于我们的 XYZLabFloor 插件,scan 法定义如下:
18 def scan(self, filename): 19 """ 20 Scans a file to check if it is supported by the plugin 21 22 @param filename : The path to be scanned. 23 @type : string 24 25 @return A list of LabFloorItems 26 """ 27 # Setup a resulting vector. 28 result = [] 29 30 # Determine extension 31 basename = os.path.basename(filename) 32 no_extension_name, extension = os.path.splitext(basename) 33 34 # Return empty string if extension isn't ".xyz" 35 if extension != '.xyz': 36 return result 37 38 # Try to load configuration 39 try: 40 reader = XYZFileRawReader(filename) 41 except Exception: 42 return result 43 44 for molecule_idx in xrange(reader.numOfMolecules()): 45 46 # Read the comment for this molecule. 47 comment = reader.comment(molecule=molecule_idx) 48 49 # Create and add LabFloorItem to list 50 if reader.numOfMolecules() == 1: 51 title = no_extension_name 52 else: 53 title = no_extension_name + " (" + str(molecule_idx) + ")" 54 55 # Create labfloor item. 56 item = LabFloorItem(MoleculeConfiguration, 57 title=title, 58 tool_tip=comment, 59 molecule_idx=molecule_idx) 60 61 # Add to result list. 62 result.append(item) 63 64 # Return the result list. 65 return result
该代码通过首先测试文件名中是否含有 “xyz” 的扩展名,然后尝试实际解析文件来检测文件是否为有效的 XYZ 文件。如果文件不是有效的 XYZ 文件,则返回空列表。如果它是有效文件,则读入文件中包含的每个分子,并为每个分子创建 LabFloorItem。
LabFloorItem 是一个 LabFloor 上表示项目的分类。它包含对象的类型,在本例中,它是 MoleculeConfiguration 以及标题和工具提示(当鼠标光标悬停在项目上时可见的文本)。有关该项目的额外信息也可以作为关键参数传递。正如我们将在下面看到的,这些参数将传递给 load 方式。
当用户与项目交互时,QuantumATK 会调用 load 方式。例如,当使用 viewer 可视化结构或将其导入 builder 时会发生这种情况。Load 法的定义为:
67 def load(self, filename, molecule_idx=0): 68 """ 69 Load the desired object in memory. 70 71 @param filename : The path of the XYZ-file. 72 @type : string 73 74 @return Desired object (MoleculeConfiguration) 75 """ 76 # Read the file 77 reader = XYZFileRawReader(filename) 78 79 # Lists of elements and positions. 80 elements = [] 81 positions = [] 82 83 # Loop over atoms. 84 for atom in reader.atomList(molecule=molecule_idx): 85 elements.append(SYMBOL_TO_ELEMENT[atom["element"]]) 86 positions.append(atom["coords"]*Angstrom) 87 88 # Create configuration. 89 configuration = MoleculeConfiguration( 90 elements=elements, 91 cartesian_coordinates=positions) 92 93 return configuration
该方式读取文件内容并提取所要求分子的元素和位置。该方法被传递了要读取分子的索引(信息存储在 scan 式中创建的 LabFloorItem 中)并创建了一个 MoleculeConfiguration。MoleculeConfiguration 类是 QuantumATK 中表示分子的方式。对于周期系统,有一个相应的 BulkConfiguration 类,用于存储晶格矢量以及坐标和元素。
此附加组件的完整源代码可在此处下载 ↓ source code。
例二:导出构型的插件
在此示例中,我们将编写一个插件以允许 QuantumATK 将结构导出到 XYZ 文件。该模块的目录结构如下所示:
XYZExporter/
__init__.py
XYZExporter.py
第一步是编译 init.py 文件:
1 import datetime 2 import XYZExporterPlugin 3 4 __addon_description__ = "Plugins for exporting XYZ files." 5 __addon_version__ = "1.0" 6 __addon_date__ = datetime.date(year=2015, month=8, day=6) 7 8 __plugins__ = [XYZExporterPlugin.XYZExporterPlugin] 9 10 def reloadPlugins(): 11 reload(XYZExporterPlugin)
下一步就是要编写真正的插件类。在之前的示例中,插件类是从 LabFloorImporterPlugin 类派生的,表明它是一个用于导入数据的插件。但本例的插件将派生自 ExportConfigurationPlugin。这些插件用于扩展 builder 支持的导出格式数量。打开构建器后,可以选择 File→Export 查看支持的文件格式列表。
从 ExportConfigurationPlugin 继承的类必须执行四种方式:title,extension,canExport 和 export。Title 式需要返回此类文件的名称 (“XYZ”)。Extension 式应该返回文件扩展名 (“xyz”)。canExport 式确定此插件是否支持结构类型(MoleculeConfiguration,BulkConfiguration,DeviceConfiguration 或 NudgedElasticBand)(XYZ文件仅支持 MoleculeConfiguration 对象)。最后,export 式是将结构传入并写进磁盘的位置。
以下为 XYZExporterPlugin.py 文件中 XYZExporterPlugin 的代码:
1 from NL.CommonConcepts.Configurations.MoleculeConfiguration import MoleculeConfiguration 2 from NL.CommonConcepts.PhysicalQuantity import Angstrom 3 4 from API import ExportConfigurationPlugin, showMessage 5 6 class XYZExporterPlugin(ExportConfigurationPlugin): 7 """ Class for handling the export of the XYZ input files. """ 8 9 def title(self): 10 """ Return the title file selection dialog. """ 11 12 return 'XYZ' 13 14 def extension(self): 15 """ The default extension of XYZ. """ 16 17 return 'xyz' 18 19 def export(self, configuration, path): 20 """ 21 Export the configuration. 22 23 @param configuration : The configuration to export. 24 @param path : The path to save the configuration to. 25 26 @return None 27 """ 28 29 # XYZ files only supports molecules. 30 if not isinstance(configuration, MoleculeConfiguration): 31 showMessage('XYZExporter can only export MoleculeConfigurations') 32 return 33 34 # Open the file with write permission. 35 with open(path, 'w') as f: 36 37 # Get the total number of atoms. 38 number_of_atoms = len(configuration) 39 40 # Write out the header to the file. 41 f.write('%i\n' % number_of_atoms) 42 f.write('Generated by XYZExporter\n') 43 44 # Get the list of atomic symbols. 45 symbols = [ element.symbol() for element in configuration.elements() ] 46 # Get the cartesian coordinates in units of Angstrom. 47 coordinates = configuration.cartesianCoordinates().inUnitsOf(Angstrom) 48 49 # Loop over each atom and write out its symbol and coordinates. 50 for i in xrange(number_of_atoms): 51 x, y, z = coordinates[i] 52 f.write('%3s %16.8f %16.8f %16.8f\n' % (symbols[i], x, y, z)) 53 54 55 def canExport(self, configuration): 56 """ 57 Method to determine if an exporter class can export a given configuration. 58 59 @param configuration : The configuration to test. 60 61 @return A bool, True if the plugin can export, False if it cannot. 62 """ 63 64 supported_configurations = [MoleculeConfiguration] 65 66 return isinstance(configuration, supported_configurations)
此附加组件的完整源代码可在此处下载 ↓ source code。
例三:读取电子密度的插件
在这个例子中,我们将创建一个读取电子密度数据的新插件。在本教程中,我们将为存储在 NumPy 二进制 .npz 文件中的 3D 网格数据定义一种新格式。
构造密度
此代码将定义电子密度并将其保存到文件 ↓ electron_density.npz。以下将是我们的插件将读取的密度文件。
1 import numpy 2 3 # Define a orthogonal cell. 4 cell = numpy.array( [ [ 5.0, 0.0, 0.0 ], 5 [ 0.0, 5.0, 0.0 ], 6 [ 0.0, 0.0, 5.0 ] ] ) 7 # Create a 3D grid of points from 0 to 5 in x, y, and z. 8 x = numpy.linspace(0.0, 5.0) 9 y = numpy.linspace(0.0, 5.0) 10 z = numpy.linspace(0.0, 5.0) 11 xx, yy, zz = numpy.meshgrid(x, y, z, indexing='ij') 12 13 # Define an electron density as a Gaussian centered at (2.5, 2.5, 2.5) times a 14 # sine wave in the x direction. 15 density = numpy.exp(-(xx-2.5)**2 - (yy-2.5)**2 - (zz-2.5)**2) * numpy.sin(yy-2.5) 16 17 # Save the cell and density to a .npz file. 18 numpy.savez('electron_density.npz', cell=cell, density=density)
这种电子密度不是物理意义上的,因为它在某些地方会是负的,但它可以让我们轻易地仔细检查数据是否被正确读取。可以在此处下载脚本 ↓ source code。
编写 NPZ 滤波器附加组件
附加组件的目录结构如下所示:
NPZFilters/
__init__.py
NPZLabFloor.py
与前面的示例一样,我们将首先关注 init.py 文件:
1 import datetime 2 import NPZLabFloor 3 4 __addon_description__ = "Plugin for reading a NPZ formatted electron density." 5 __addon_version__ = "1.0" 6 __addon_date__ = datetime.date(2014, 9, 1) 7 8 __plugins__ = [NPZLabFloor.NPZLabFloor] 9 10 def reloadPlugins(): 11 reload(NPZLabFloor)
这里没有什么新的内容。现在我们需要定义实际的插件类:
1 import numpy 2 import os 3 4 import NLEngine 5 6 from API import LabFloorImporterPlugin 7 from API import LabFloorItem 8 from NL.Analysis.ElectronDensity import ElectronDensity 9 from NL.Analysis.GridValues import GridValues 10 from NL.ComputerScienceUtilities.NLFlag import Spin 11 from NL.CommonConcepts.PhysicalQuantity import Angstrom 12 13class NPZLabFloor(LabFloorImporterPlugin): 14 """ 15 Class for handling the importing of NPZ-files as LabFloor items. 16 """ 17 18 def scan(self, filename): 19 """ 20 Scans a file to check if it is supported by the plugin 21 22 @param filename : The path to be scanned. 23 @type : string 24 25 @return A list of LabFloorItems 26 """ 27 # Setup a vector for the LabFloorItems that will be returned. 28 lab_floor_items = [] 29 30 # Determine extension 31 basename = os.path.basename(filename) 32 no_extension_name, extension = os.path.splitext(basename) 33 34 # Return an empty list if the extension isn't ".npz" 35 if extension != '.npz': 36 return [] 37 38 item = LabFloorItem(ElectronDensity, 39 title='NPZ Electron Density', 40 tool_tip='NPZ Electron Density') 41 42 # Add to the list of items. 43 lab_floor_items.append(item) 44 45 # Return the list of items. 46 return lab_floor_items 47 48 def load(self, filename): 49 """ 50 Load the desired object in memory. 51 52 @param filename : The path of the NPZ-file. 53 @type : string 54 55 @return Desired object (MoleculeConfiguration) 56 """ 57 58 # Read the file 59 npz = numpy.load(filename) 60 61 # Create an "empty" ElectronDensity object. 62 electron_density = ElectronDensity.__new__(ElectronDensity) 63 64 # We will now fill out a dictionary that contains the information 65 # needed by the ElectronDensity class. 66 data = {} 67 68 # The "data" key is the electron density. The units must be given in the 69 # "data_unit" key. The array should have have the x-axis as the first 70 # dimension, the y-axis as the second, and the z-axis as the third. 71 data['data'] = npz['density'] 72 73 # The data in "data" has no units so they are assigned here. 74 data['data_unit'] = 1.0 * Angstrom**-3 75 76 # Set the origin to be at zero. 77 data['origo'] = numpy.zeros(3) 78 79 # The cell must be given in Bohr units. 80 data['cell'] = npz['cell'] 81 82 # The boundary conditions are expressed as a list of 6 numbers that should 83 # map to: 84 # { Dirichlet, Neumann, Periodic, Multipole }; 85 # A value of 2 corresponds to "Periodic". 86 data['boundary_conditions'] = [2, 2, 2, 2, 2, 2] 87 88 # Construct the GridValues specific part of the object. 89 GridValues._populateFromDict(electron_density, data) 90 91 # Set the spin_type to unpolarized. 92 spin_type = NLEngine.UNPOLARIZED 93 electron_density._AnalysisSpin__spin_type = spin_type 94 95 sp = Spin.All 96 electron_density._AnalysisSpin__spin = sp 97 electron_density._setSupportedSpins(sp) 98 99 return electron_density
这个附加组件的完整代码可在此处下载 ↓ source code。
如何安装附加组件
有两种不同的安装附加组件的方法。第一种,设置环境变量 QUANTUM_ADDONS_PATH 到插件模块所在的目录。例如,在先前的章节中 NPZFilters 附件的路径为 $HOME/AddOns/NPZFilters,然后设置环境变量为 QUANTUM_ADDONS_PATH=$HOME/AddOns 即可。
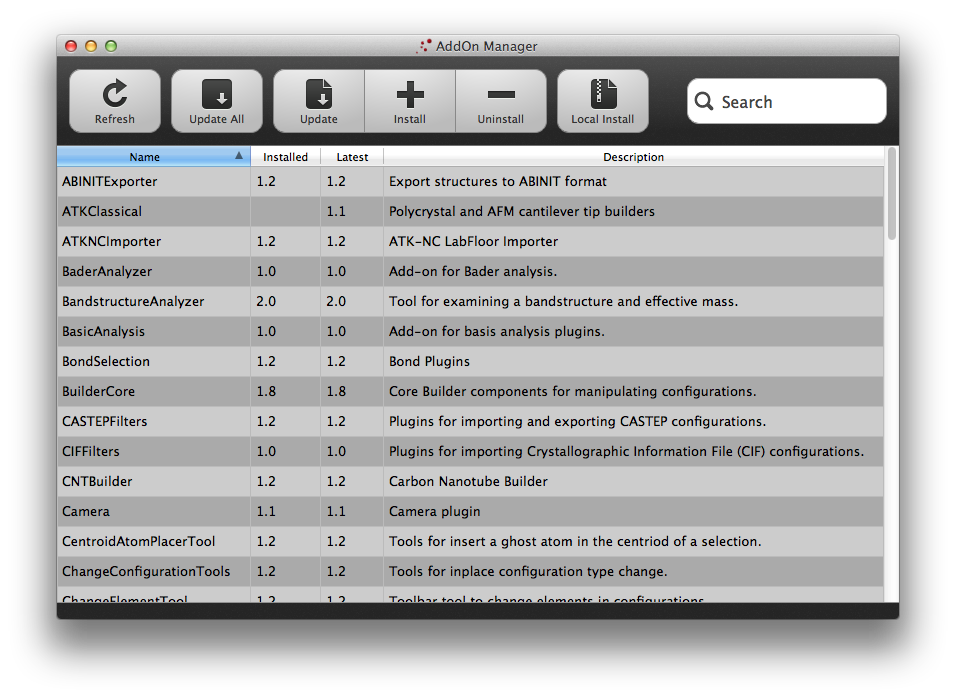
另一种方法是压缩 Python 模块并通过图形界面安装。第一步是创建一个包含该模块的 zip 文件。按照上一段中的 NPZFilter 示例,可以通过运行 zip -r NPZFilters.zip $ HOME / AddOns / NPZFilters 来完成。然后,在 QuantumATK 中,Help 菜单下面有一个名为 AddOn Manager 的选项。打开 AddOn Manager 将会呈现出如下所示的窗口:
点击 Local Install,出现一个文件对话框。选择 NPZFilters.zip,QuantumATK 将安装插件到 QUANTUM_ADDONS_PATH。
测试 NPZ 滤波器附加组件
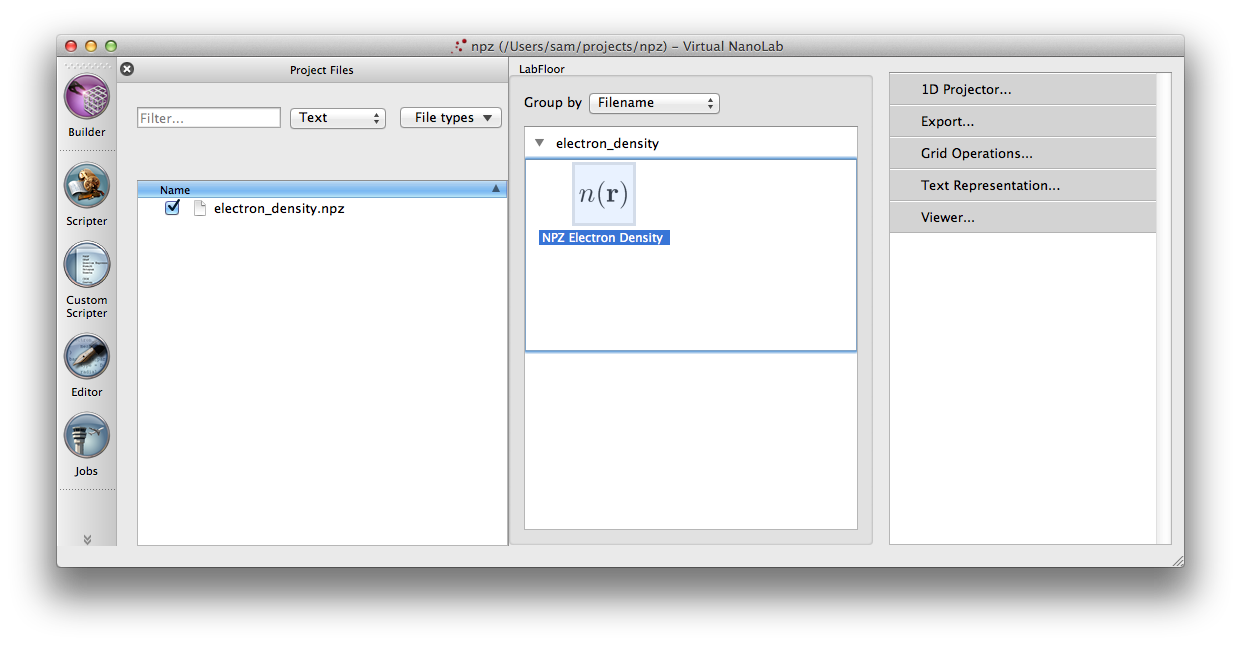
按照上一节中的步骤操作后,现在应该已经安装上了 NPZFilters AddOn。您可以通过拉出 AddOn Manager 并查看列表中的 NPZFilters 再次确认已安装。如果我们在 QuantumATK 中创建一个新项目,且该文件夹包含我们创建的 electron_density.npz 文件,那么 ElectronDensity 对象就应该显示在 lab floor 上。
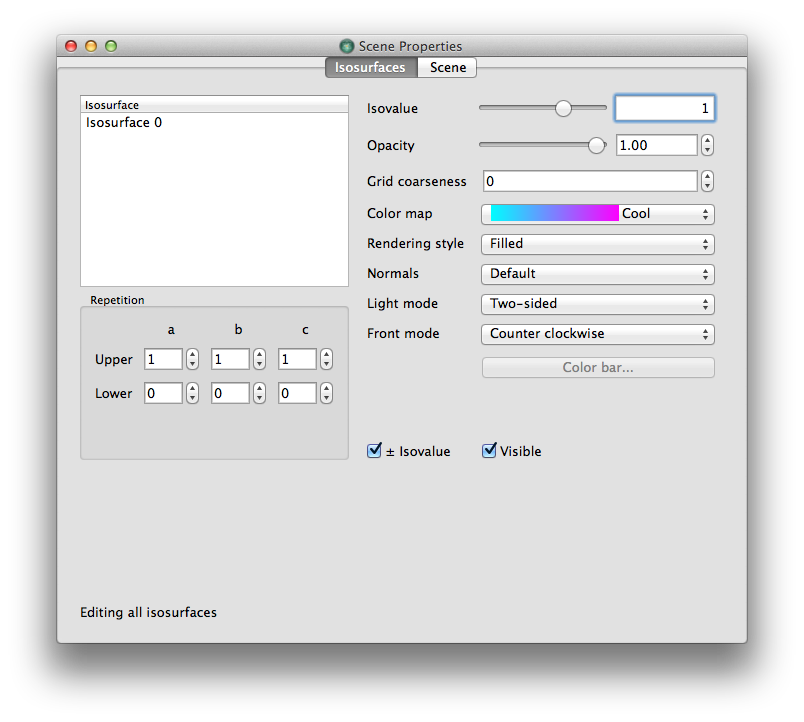
点击右侧面板上的 Viewer… 按钮将电子密度可视化。在出现的对话框中,有 isosurface 和 cut plane 可供选择。选择 isosurface。默认的 isosurface 值是平均电荷密度,对于我们的电荷密度来说为零(因为一半是负,一半是正)。单击右侧面板中的 Properties … 按钮,将 Isovalue … 滑块拖动到接近 1 的地方。
得到的等值面现在应该看起来像哑铃,两种不同的颜色分别代表负密度和正密度的区域,以及通过 x-z 轴的零密度平面。这证实了我们在模型密度函数中的读取结果正确。